Kit de isolamento de RNA total vegetal mais kit de purificação de RNA total para plantas ricas em polissacarídeos e polifenóis
Especificações
50 preparações, 200 preparações
O kit usa a coluna giratória e a fórmula desenvolvida pela Foregene, que pode extrair eficientemente RNA total de alta pureza e alta qualidade de vários tecidos vegetais com alto teor de polissacarídeos ou polifenóis.Ele fornece a coluna de limpeza de DNA que pode remover facilmente o DNA genômico do sobrenadante e do tecido lisado.A coluna somente de RNA pode efetivamente ligar o RNA.O kit pode processar um grande número de amostras ao mesmo tempo.
Todo o sistema não contém RNase, portanto o RNA purificado não será degradado.Buffer PRW1 e Buffer PRW2 podem garantir que o RNA obtido não seja contaminado por proteínas, DNA, íons e compostos orgânicos.
Componentes do kit
| Buffer PSL1, Buffer PS, Buffer PSL2 |
| Tampão PRW1, Tampão PRW2 |
| RNase-Free ddH2O, coluna de limpeza de DNA |
| Coluna Somente de RNA |
Características&vantagens
■ Operação em temperatura ambiente (15-25℃) durante todo o processo, sem banho de gelo e centrifugação em baixa temperatura.
■ Kit completo RNase-Free, não precisa se preocupar com a degradação do RNA.
■ Particularmente adequado para a purificação de RNA de amostras de plantas de polissacarídeos e polifenóis.
■ A coluna de limpeza de DNA liga-se especificamente ao DNA, para que o kit possa remover a contaminação do DNA genômico sem adicionar DNase.
■ Alto rendimento de RNA: a coluna somente de RNA e a fórmula exclusiva podem purificar o RNA com eficiência.
■ Velocidade rápida: fácil de operar e pode ser concluída em 30 minutos.
■ Segurança: nenhum reagente orgânico é necessário.
■ Alta qualidade: Os fragmentos de RNA purificados são de alta pureza, livres de proteínas e outras impurezas e podem atender a várias aplicações experimentais downstream.
Parâmetros do produto
■ Aplicações downstream: síntese de cDNA de primeira cadeia, RT-PCR, clonagem molecular, Northern Blot, etc.
■ Amostra: Tecidos vegetais frescos ou congelados de polissacarídeos e polifenóis
■ Dosagem: 50mg de tecido vegetal
■ Capacidade máxima de ligação de RNA da coluna de purificação: 80 μg
■ Volume de eluição: 50-200 μl
Kit de aplicação
É adequado para a extração e purificação de RNA total de amostras de tecidos de plantas frescas ou congeladas (especialmente tecido de folhas de plantas frescas) com alto teor de polissacarídeos e polifenóis.
fluxo de trabalho
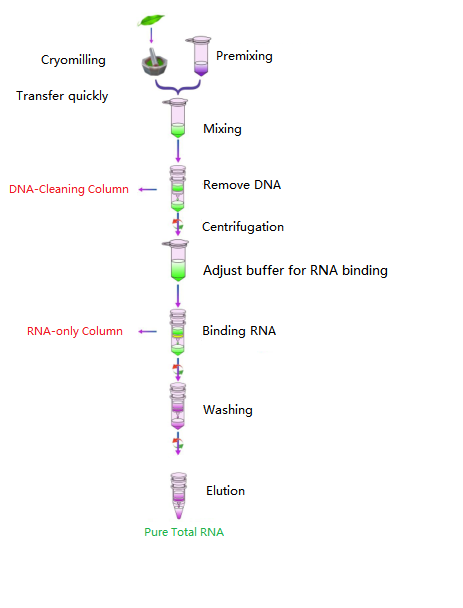
Diagrama
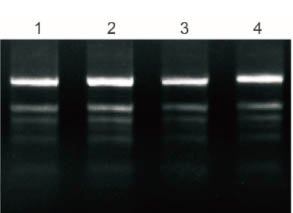
Plant Total RNA Isolation Kit Plus processou 50mg de folhas frescas de polissacarídeos e polifenóis, e 5% de RNA purificado foi testado por eletroforese.
1: Banana
2: Ginkgo
3: Algodão
4: Romã
Armazenamento e prazo de validade
Este kit pode ser armazenado por 24 meses em condições secas à temperatura ambiente (15-25 ℃);se precisar ser armazenado por mais tempo, pode ser armazenado em 2–8 ℃.
O tampão PSL1 pode ser colocado a 4 ℃ por 1 mês após a adição de β-mercaptoetanol (recomenda-se adicioná-lo ao mesmo tempo do experimento).
A coluna obstruída
Após o plugue da coluna, o rendimento do RNA é reduzido ou mesmo impossível de purificar o RNA, e a massa de RNA obtida é baixa.
Análise de causa comum:
1. As quebras de amostra não são completas.
A quebra da amostra não causa o bloqueio completo da COLUNA DE LIMPEZA DE DNA, mas afeta o rendimento e a qualidade do RNA.Recomendamos a operação de moagem rápida em nitrogênio líquido suficiente quando você quebrou as amostras. Tente esmagar a parede celular da amostra, a membrana celular e outros tecidos.Para amostras de plantas de poliol polissacarídeos, recomendamos que você use Plant Total RNA ISOLATION KIT PLUS.
2. Ao aspirar o sobrenadante da amostra separada com a coluna de limpeza de DNA, o possível precipitado fragmentado celular pode ser inalado.
Os sedimentos fragmentados de células retirados causarão a Coluna SOMENTE de RNA, que será bloqueada quando a operação de adsorção de RNA for realizada (consulte a etapa 6).Recomendamos que você tenha cuidado ao aspirar este sobrenadante para evitar que os detritos celulares sejam sugados.
3. A quantidade inicial da amostra é muito grande.
O uso excessivo de amostras resultará em fragmentação incompleta da amostra ou lise celular incompleta pelo Buffer PSL1, resultando no bloqueio da coluna de purificação durante a purificação.Plant Total RNA Isolation Kit Cada amostra operacional purificada é de 50 mg.Para amostras de plantas de poliol polissacarídeos, recomendamos que você experimente Plant Total RNA ISOLATION KIT PLUS.
4. A temperatura da centrífuga está muito baixa.
Todo o processo de isolamento e purificação do RNA é realizado em temperatura ambiente (20-25°C), exceto que o tecido da amostra é quebrado por nitrogênio líquido. A temperatura de algumas centrífugas criogênicas é inferior a 20℃, o que pode causar bloqueio da coluna de limpeza de DNA e/ou da coluna somente de RNA.Se isso acontecer, defina a temperatura da centrífuga para 20-25℃, ecertifique-se de que a mistura de lise e/ou o sobrenadante adicionado de etanol foi pré-aquecido a 37°C.
Nenhum RNA extraído ou o rendimento do RNA é baixo
Geralmente, existem muitos fatores que afetam a eficiência da recuperação, como: conteúdo de RNA da amostra, método de operação, volume de eluição, etc.
Análise de causas comuns como abaixo:
1.Um banho de gelo ou centrifugação a baixa temperatura (4°C) foi realizado durante a operação.
Sugestão: Operar em temperatura ambiente (15-25°C) em todo o processo não fazer banho de gelo e centrifugação em baixa temperatura.
2.O RNA foi degradado devido à preservação inadequada da amostra ou preservação de longo prazo da amostra.
Recomendação: Amostras coletadas recentemente devem ser rapidamente congeladas em nitrogênio líquido e, em seguida, armazenadas a -80°C por um longo período de tempo, evitar congelamento e descongelamento repetidos de amostras;ou mergulhe imediatamente as amostras em solução estabilizadora de RNA RNAlater (amostras de animais).
3. Fragmentação e lise insuficientes da amostra levam ao bloqueio da coluna de purificação.
Sugestão: Ao moer o tecido, certifique-se de que o tecido esteja suficientemente moído e transfira-o rapidamente para o Buffer PSL1 pré-preparado (confirme se a proporção correta de β-ME foi adicionada, consulte a etapa 1 do procedimento).
4.O eluente foi adicionado incorretamente.
Sugestão: Certifique-se de que o ddH2O livre de RNase seja pingado no meio da membrana da coluna de purificação.
5. O volume correto de etanol absoluto não foi adicionado ao tampão PSL2 ou tampão PRW2.
Sugestão: Siga as instruções, adicione o volume correto de etanol absoluto ao Buffer PSL2 e ao Buffer PRW2 e misture bem antes de usar o kit.
6. A quantidade de amostra de tecido é inadequada.
Sugestão: Utilizar 50 mg de tecido por 500 μl de Buffer PSL1.Usar muito tecido reduzirá a quantidade de RNA extraído e a pureza do RNA resultante também será reduzida.Recomendamos fortemente que a dosagem inicial da amostra não exceda 50 mg por operação de extração de RNA.
7.Volume de eluição inadequado ou eluição incompleta.
Sugestão: O volume do eluente da coluna de purificação é de 50-200 μl;se o efeito de eluição não for satisfatório, recomenda-se estender o tempo à temperatura ambiente após adicionar ddH2O livre de RNase pré-aquecido, como 5-10min.
8.A coluna de purificação tem resíduo de etanol após a lavagem com BufferPRW2.
Sugestão: Caso o tubo vazio seja centrifugado por 1 min e ainda reste etanol após a lavagem em Buffer PRW2, pode-se aumentar o tempo de centrifugação do tubo vazio para 2 min, ou colocar a coluna de purificação em temperatura ambiente por 5 min para remover totalmente o etanol residual.
9.O kit foi usado incorretamente.
Sugestão: Para amostras vegetais de polissacarídeos polifenólicos, o uso de kits comuns, como o Plant Total RNA Isolation Kit, pode não ser capaz de obter amostras de RNA ideais.Recomendamos que você use Plant Total RNA IsolationKit Plus, que é especialmente projetado para amostras de plantas de polissacarídeos polifenólicos.Um kit especialmente desenvolvido para extração de RNA de amostras vegetais de polifenóis e polissacarídeos.
O valor OD260/OD280 é baixo
A eluição de RNA com ddH2O e usada para leituras de espectrofotômetro resulta em valores baixos de OD260/OD280.Recomendamos o uso de Tris-HCl 10 mM, pH 7,5 (em vez de ddH2O livre de RNase para eluir o RNA) para obter valores OD260/OD280 relativamente corretos, consulte “Ensaios de concentração e purificação de RNA” na página 19.
O RNA purificado é degradado
A qualidade do RNA purificado está relacionada a fatores como preservação da amostra, contaminação por RNase e manipulação.
Análise das causas comuns:
1.As amostras de tecido não foram armazenadas a tempo após a coleta.
Recomendação: Se as amostras de tecido não forem usadas a tempo após a coleta, armazene-as imediatamente em nitrogênio líquido em baixa temperatura ou transfira-as para -80°C para armazenamento de longo prazo após congelamento rápido em nitrogênio líquido ou mergulhe imediatamente as amostras em solução estabilizadora de RNA RNAlater (amostras de animais).Para extração de RNA, tente usar amostras de tecido recém-coletadas.
2. Congelamento e descongelamento repetidos de amostras de tecido.
Sugestão: Ao armazenar amostras de tecido, é melhor cortá-las em pedaços pequenos para preservação e retirar uma parte delas ao usá-las para evitar a degradação do RNA causada pelo congelamento e descongelamento repetidos das amostras.
3.RNase é introduzido na sala de operação ou não usa luvas descartáveis, máscaras, etc.
Sugestão: Os experimentos de extração de RNA são melhor realizados em operações de RNA separadas, e a mesa do laboratório deve ser limpa antes do experimento, e luvas e máscaras descartáveis devem ser usadas durante o experimento para evitar a degradação do RNA causada pela introdução de RNase ao máximo.
4.O reagente é contaminado por RNase durante o uso.
Sugestão: Substitua por uma nova série de kits de extração de RNA total de plantas para experimentos relacionados.
5.Os tubos de centrífuga e as pontas de pipeta usados para manipulação de RNA estão contaminados com RNase.
Sugestão: Certifique-se de que os tubos de centrífuga, pontas de pipeta, pipetas, etc. usados na extração de RNA são todos RNase-Free.
Manual de instruções:
Manual de Instruções do Kit de Isolamento Total de RNA da Planta Plus