É bem conhecido que no dogma central, o RNA é o mediador transcricional entre o DNA e a expressão da proteína.Comparada com a detecção de DNA, a detecção de RNA pode refletir mais objetivamente a expressão gênica em organismos.Experimentos envolvendo RNA incluem: qRT-PCR, RNA-Seq e detecção de genes de fusão, etc.Entra lixo, sai lixo, se a qualidade do RNA não é boa, então os resultados experimentais devem ser insatisfatórios, manifestados especificamente como dados imprecisos ou baixa repetibilidade.Portanto, mais atenção deve ser dada ao processamento do RNA, e o link de controle de qualidade também é mais importante para garantir a precisão e exatidão dos dados experimentais subsequentes.
Para o controle de qualidade do RNA, geralmente existem os seguintes métodos comumente usados:
- Espectrofotometria
- eletroforese em gel de agarose
- Bioanalisador Agilent
- PCR quantitativo fluorescente em tempo real
- Método de corante fluorescente Qubit
01 Espectrofotometria
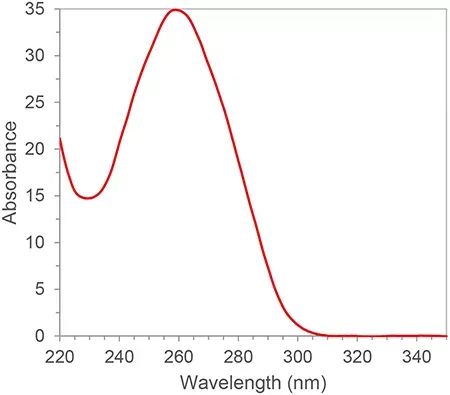
O RNA possui ligações duplas conjugadas e tem um pico de absorção em um comprimento de onda de 260 nm.De acordo com a lei de Lambert-Beer, podemos calcular a concentração de RNA a partir do pico de absorção em 260nm.Além disso, também podemos calcular a pureza do RNA de acordo com a proporção de picos de absorção de 260nm, 280nm e 230nm.280nm e 230nm são os picos de absorção de proteínas e pequenas moléculas, respectivamente.A proporção de A260/A280 e A260/A230 de pureza de RNA qualificada deve ser maior que 2. Se for menor que 2, significa que há contaminação por proteína ou molécula pequena na amostra de RNA e precisa ser purificada novamente.As fontes de contaminação afetarão os experimentos a jusante, como a inibição da eficiência de amplificação das reações de PCR, resultando em resultados quantitativos imprecisos.A pureza do RNA tem grande influência nos resultados subsequentes, então a espectrofotometria é geralmente um elo de controle de qualidade indispensável na primeira etapa dos experimentos com ácidos nucleicos.
Figura 1. Espectro Típico de Absorção de RNA/DNA
02 Eletroforese em gel de agarose
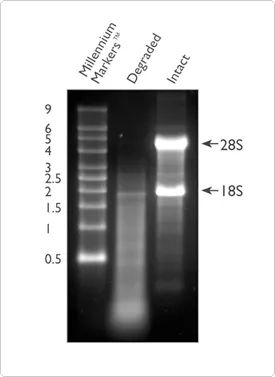
Além da pureza, a integridade do RNA também é um dos indicadores importantes para julgar a qualidade do RNA.A degradação do RNA levará a um grande número de fragmentos curtos na amostra, de modo que o número de fragmentos de RNA que podem ser efetivamente detectados e cobertos pela sequência de referência será reduzido.A integridade do RNA pode ser verificada por eletroforese do RNA total em um gel de agarose a 1%.Este método pode configurar o gel você mesmo ou usar o sistema E-Gel™ pré-fabricado para teste de integridade.Mais de 80% do RNA total é RNA ribossômico, a maioria dos quais é composta por rRNA 28S e 18S (em sistemas de mamíferos).O RNA de boa qualidade mostrará duas barras brilhantes óbvias, que são barras brilhantes 28S e 18S, respectivamente, a 5 Kb e 2 Kb, e a proporção tenderá a ser próxima de 2:1.Se estiver em estado difuso, significa que a amostra de RNA pode ter sido degradada e é recomendável usar o método descrito posteriormente para testar ainda mais a qualidade do RNA.
Figura 2. Comparação de RNA degradado (pista 2) e intacto (pista 3) na eletroforese em gel de agarose
03 Bioanalisador Agilent
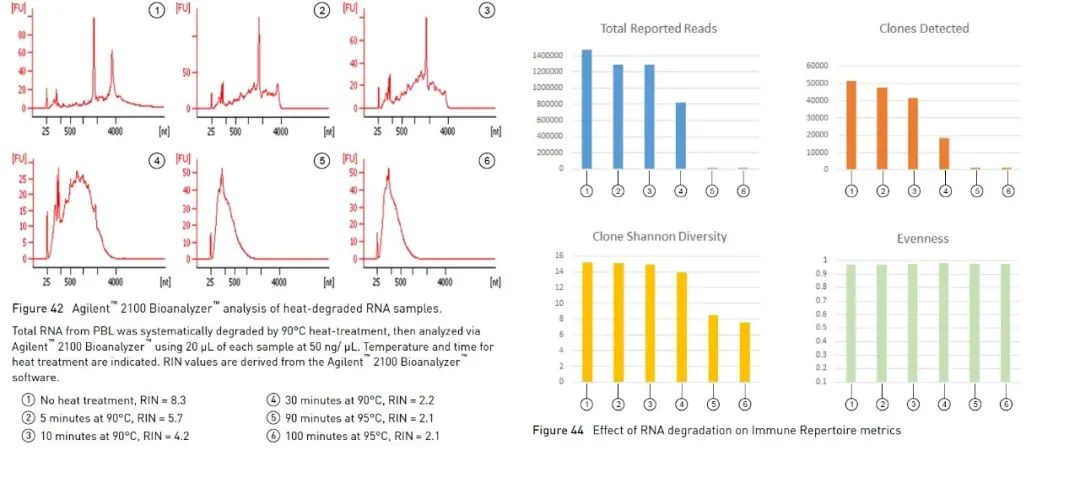
Além do método de eletroforese em gel de agarose descrito acima, que pode nos ajudar a identificar a integridade do RNA de forma simples e rápida, também podemos usar o bioanalisador Agilent para determinar a integridade do RNA.Ele usa uma combinação de microfluídica, eletroforese capilar e fluorescência para avaliar a concentração e integridade do RNA.Usando o algoritmo integrado para analisar o perfil da amostra de RNA, o bioanalisador Agilent pode calcular um valor de integridade de RNA de referência, Número de Integridade de RNA (doravante referido como RIN) [1].Quanto maior o valor de RIN, maior a integridade do RNA (1 é extremamente degradado, 10 é o mais completo).Alguns experimentos envolvendo RNA sugerem o uso do RIN como parâmetro para avaliação de qualidade.Tomando experimentos de sequenciamento de alto rendimento (doravante referidos como NGS) como exemplo, as diretrizes do Oncomine™ Human Immune Repertoire, que é usado para detectar receptores de antígenos de células B e T na série de painéis Oncomine da Thermo Fisher, sugerem que amostras com valores de RIN superiores a 4, leituras e clones mais eficazes podem ser medidos (Figura 3).Existem diferentes intervalos recomendados para diferentes painéis e, muitas vezes, um RIN mais alto pode trazer dados mais eficazes.
Figura 3, em experimentos do Oncomine™ Human Immune Repertoire, amostras com RIN maior que 4 podem detectar leituras mais efetivas e clones de células T.【2】
No entanto, o valor RIN também possui algumas limitações.Embora o RIN tenha uma alta correlação com a qualidade dos dados experimentais do NGS, ele não é adequado para amostras FFPE.As amostras de FFPE foram tratadas quimicamente por um longo tempo, e o RNA extraído geralmente tem um valor de RIN relativamente baixo.No entanto, isso não significa que os dados efetivos do experimento devam ser insatisfatórios.Para avaliar com precisão a qualidade das amostras FFPE, precisamos usar medições diferentes do RIN.Além do RIN, o bioanalisador Agilent também pode calcular o valor DV200 como um parâmetro de avaliação da qualidade do RNA.DV200 é um parâmetro que calcula a proporção de fragmentos maiores que 200 pb em uma amostra de RNA.O DV200 é um melhor indicador da qualidade da amostra FFPE do que o RIN.Para o RNA extraído pelo FFPE, tem uma correlação muito alta com o número de genes que podem ser efetivamente detectados e a diversidade de genes [3].Embora o DV200 possa compensar as deficiências na detecção de qualidade do FFPE, o bioanalisador Agilent ainda não pode analisar de forma abrangente os problemas de qualidade em amostras de RNA, incluindo se há inibidores nas amostras.Os próprios inibidores podem afetar a eficiência de amplificação de experimentos downstream e reduzir a quantidade de dados úteis.Para saber se há um inibidor na amostra, podemos adotar o método de PCR quantitativo fluorescente em tempo real descrito a seguir.
04 PCR quantitativo fluorescente em tempo real
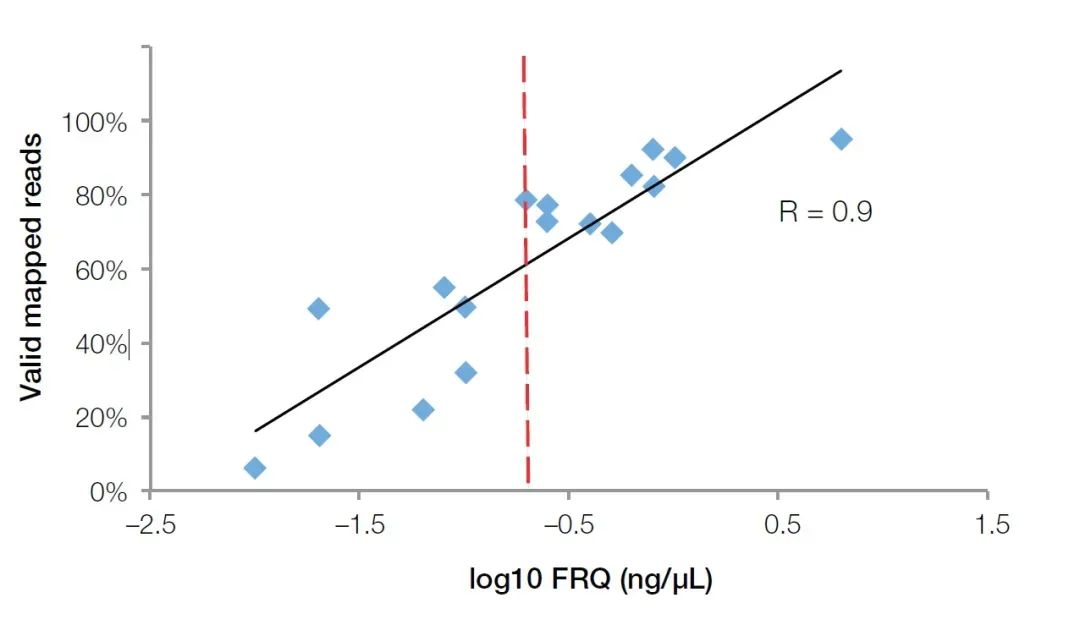
O método PCR quantitativo fluorescente em tempo real pode não apenas detectar os inibidores na amostra, mas também refletir com precisão a qualidade do RNA na amostra FFPE.Em comparação com os analisadores biológicos Agilent, os instrumentos quantitativos de fluorescência em tempo real são mais populares nos principais laboratórios biológicos devido à sua aplicação mais ampla.Para testar a qualidade das amostras de RNA, basta adquirir ou preparar sondas de primer para genes de referência internos, como o GUSB (Cat n.º Hs00939627).Ao usar este conjunto de primers, sondas e padrões (RNA total de concentração conhecida) para realizar experimentos quantitativos absolutos, a concentração efetiva do fragmento de RNA pode ser calculada como o padrão de avaliação da qualidade do RNA (Quantificação Funcional de RNA (FRQ) para abreviar).Em um teste NGS, descobrimos que o FRQ das amostras de RNA tem uma correlação muito alta com o volume de dados efetivo.Para todas as amostras maiores que 0,2 ng/uL FRQ, pelo menos 70% das leituras podem efetivamente cobrir a sequência de referência (Figura 4).
Na Figura 4, o valor de FRQ detectado pelo método quantitativo de fluorescência tem uma correlação muito alta (R2>0,9) com os dados efetivos obtidos no experimento NGS.A linha vermelha é o valor de FRQ igual a 0,2 ng/uL (log10 = -0,7).【4】
Além de ser aplicável a amostras FFPE, o método de PCR quantitativo em tempo real também pode monitorar efetivamente os inibidores nas amostras.Podemos adicionar a amostra a ser detectada no sistema de reação com Controle Positivo Interno (IPC) e seu Ensaio, e então realizar a quantificação de fluorescência para obter o valor de Ct.Se o valor Ct ficar atrás do valor Ct na reação sem amostra, isso indica que o inibidor está presente na amostra e inibe a eficiência da amplificação na reação.
05 método de corante fluorescente Qubit
Qubit Fluorometer é o pequeno dispositivo mais comumente usado para concentração de ácido nucleico e detecção de pureza, que é fácil de operar e existe em quase todos os laboratórios de biologia molecular.Ele calcula com precisão a concentração de ácido nucleico detectando um corante fluorescente de ligação de ácido nucleico (reagente de detecção Qubit).O Qubit tem alta sensibilidade e especificidade e pode quantificar com precisão o RNA até a concentração de pg/µL.Além da conhecida capacidade de quantificar com precisão a concentração de ácido nucleico, o novo modelo mais recente da Thermo Fisher, o Qubit 4.0, também pode detectar a integridade do RNA.O sistema de detecção de RNA do Qubit 4.0 (RNA IQ Assay) detecta a integridade do RNA detectando simultaneamente dois corantes fluorescentes específicos.Esses dois corantes fluorescentes podem se ligar a grandes fragmentos e pequenos fragmentos de RNA, respectivamente.Esses dois corantes fluorescentes indicam a proporção de grandes fragmentos de RNA na amostra e, a partir disso, o valor de QI (Integridade e Qualidade) que representa a qualidade do RNA pode ser calculado.O valor de QI é aplicável a amostras FFPE e não FFPE e tem grande influência na qualidade do sequenciamento subsequente.Tomando como exemplo os experimentos NGS, nos experimentos de teste RNA-Seq realizados na plataforma Ion torrent™, a maioria das amostras com valores de QI superiores a 4 tiveram pelo menos 50% de leituras efetivas (Figura 5).Comparado com os métodos de detecção mencionados acima, o Qubit IQ Assay não é apenas mais conveniente de operar e leva menos tempo (em cinco minutos), mas também tem uma grande correlação entre o valor medido do parâmetro IQ e a qualidade dos dados dos experimentos a jusante.
Na Figura 5, há uma grande correlação entre o valor do Qubit RNA IQ e as leituras mapeadas do RNA-Seq.【5】
Através da introdução acima, acredito que todos tenham uma compreensão suficiente dos diferentes métodos de controle de qualidade do RNA.Na prática, você pode escolher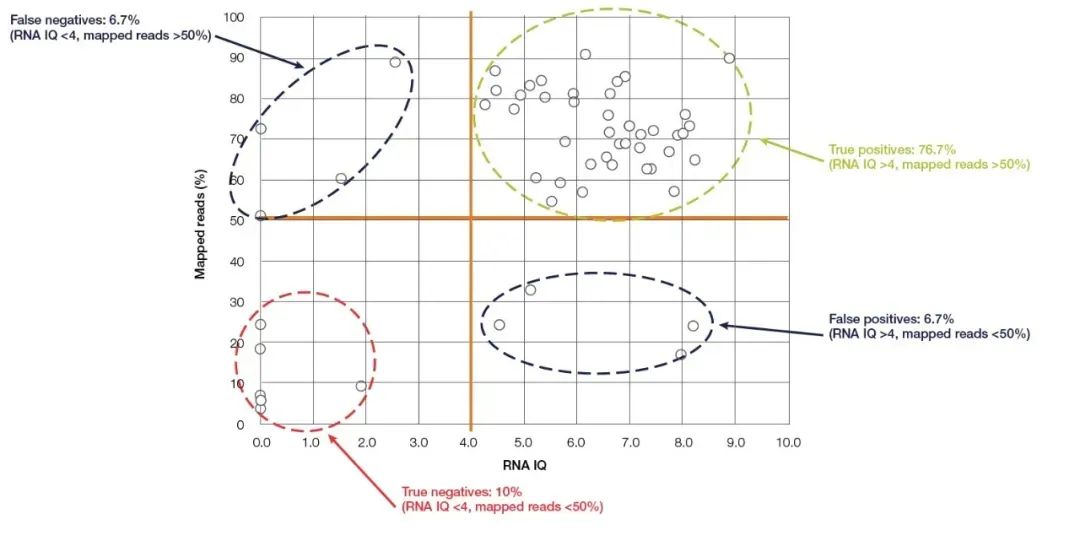 o método correspondente de acordo com o tipo de amostra e instrumentos existentes.Somente controlando bem a qualidade do RNA podemos evitar o fracasso de experimentos subsequentes causados pela baixa qualidade da amostra, economizando tempo, energia e custos preciosos.
o método correspondente de acordo com o tipo de amostra e instrumentos existentes.Somente controlando bem a qualidade do RNA podemos evitar o fracasso de experimentos subsequentes causados pela baixa qualidade da amostra, economizando tempo, energia e custos preciosos.
Produtos de referência:
Kit de isolamento de RNA total animal
Kit de isolamento de RNA total da célula
referências
【1】Schroeder, A., Mueller, O., Stocker, S. et al.O RIN: um número de integridade de RNA para atribuir valores de integridade a medições de RNA.BMC Molecular Biol 7, 3 (2006).https://doi.org/10.1186/1471-21 99-7-3
【2】Guia do Usuário do Repertório Imune Humano Oncomine (Pub. No. MAN0017438 Rev. C.0).
【3】Leah C Wehmas, Charles E Wood, Brian N Chorley, Carole L Yauk, Gail M Nelson, Susan D Hester, Métricas de qualidade aprimoradas para avaliar o RNA derivado de amostras de tecido embebidas em parafina fixadas em formalina arquivísticas, Ciências toxicológicas, Volume 170, Edição 2, agosto de 2019, páginas 357–373,https://doi.org/10.1093/toxsci/
Horário da postagem: 12 de junho de 2023