Especificidade da detecção
Na maioria dos casos, o objetivo do design do primer é maximizar a especificidade da PCR.Isso é determinado pela influência mais ou menos previsível de muitas variáveis.Uma variável importante é a sequência na extremidade 3' do iniciador.
É importante ressaltar que os ensaios de PCR projetados para especificidade têm maior probabilidade de manter alta eficiência em uma ampla faixa dinâmica, porque o ensaio não produz produtos de amplificação não específicos, competindo com os reagentes de PCR ou inibindo a principal reação de amplificação.
É claro que, em alguns casos, a especificidade não é o mais importante, por exemplo, quando o objetivo é quantificar patógenos intimamente relacionados, mas diferentes patógenos, design especial, padrões de otimização e verificação são necessários.
A curva de fusão é um método padrão para avaliar a especificidade dos amplicons, pelo menos em termos de amplificar um único alvo.No entanto, deve-se enfatizar que as curvas de fusão podem ser enganosas porque, por exemplo, podem ser afetadas pelos efeitos combinados de primers subótimos e baixas concentrações de molde.
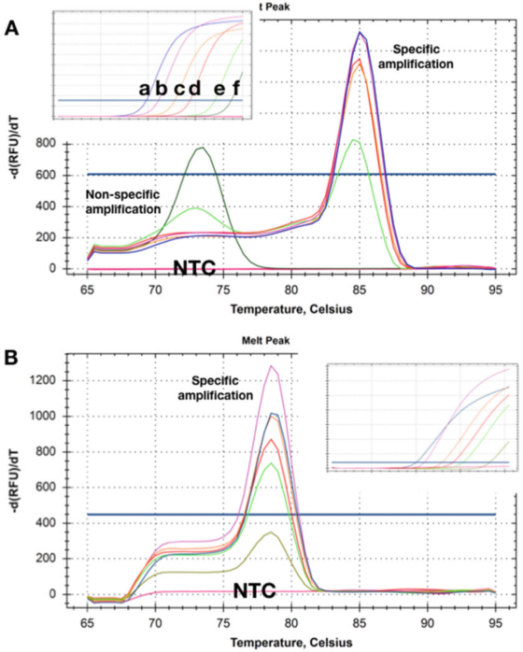
P5 |A curva de fusão mostra os desvios de Tm obtidos a partir de duas detecções de diferentes quantidades de dois DNAs alvo.
A. Em concentrações mais altas (ad)), não há nenhum dímero de primer óbvio após a conclusão da medição qPCR.À medida que a concentração do molde diminui para 50 cópias (e), um produto não específico começa a aparecer e se torna o único produto na concentração mais baixa (f).
B. O teste registrou o mesmo Tms em todas as concentrações alvo, e não houve nenhum dímero de primer óbvio mesmo na concentração mais baixa (5 cópias).Ao usar esses dois métodos de detecção, nenhum produto de amplificação foi detectado nos NTCs.
P5 mostra as curvas de dissolução obtidas com amostras nas quais o molde está presente em diferentes concentrações.P 5a mostra que nas duas concentrações mais baixas, os Tms dos produtos de amplificação não específicos produzidos são inferiores aos dos amplicões específicos.
Obviamente, este método de detecção não pode ser usado de forma confiável para detectar alvos que existem em baixas concentrações.
Curiosamente, os NTCs, ou seja, amostras sem nenhum DNA, não registraram produtos de amplificação (não específicos), indicando que o DNA genômico de fundo pode participar da amplificação/polimerização não específica.
Às vezes, esses primers de fundo e amplificação não específica não podem ser remediados, mas muitas vezes é possível projetar um método de detecção que não tenha amplificação não específica em qualquer concentração de modelo e NTC (P 5b).
Aqui, mesmo registrando a amplificação da concentração alvo com um Cq de 35 produzirá uma curva de dissolução específica.Da mesma forma, os NTCs não mostraram sinais de amplificação inespecífica.Às vezes, o comportamento de detecção pode ser dependente do licor-mãe, e apenas amplificação não específica é detectada em certas composições de tampão, que podem estar relacionadas a diferentes concentrações de Mg2+.
Estabilidade de detecção
A otimização de Ta é uma etapa útil no processo de verificação empírica e otimização da detecção de qPCR.Ele fornece uma indicação direta da robustez do conjunto de primers, mostrando a temperatura (ou faixa de temperatura) que produz o Cq mais baixo sem amplificar o NTC.
A diferença de duas a quatro vezes na sensibilidade pode não ser importante para pessoas com alta expressão de mRNA, mas para testes de diagnóstico, pode significar a diferença entre resultados positivos e falsos negativos.
As propriedades Ta dos primers qPCR podem variar muito.Alguns testes não são muito robustos e, se não forem realizados sob o valor de Ta ideal dos primers, eles entrarão em colapso rapidamente.
Isso é importante porque esse tipo de detecção costuma ser problemático no mundo real, e a pureza da amostra, a concentração de DNA ou a presença de outro DNA podem não ser ideais.
Além disso, o número de cópias de destino pode variar em uma ampla faixa e os reagentes, utensílios de plástico ou instrumentos podem ser diferentes daqueles usados na configuração do teste.
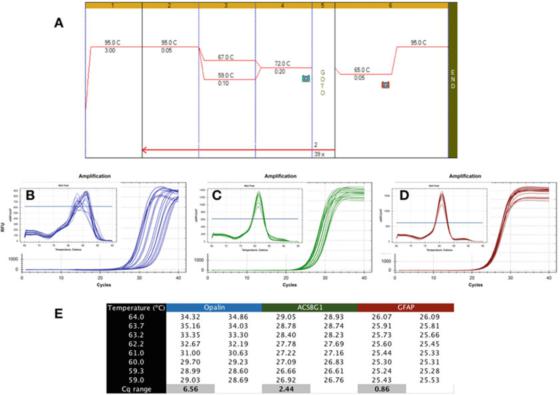
P6|O gradiente de temperatura mostra a robustez diferente da detecção de PCR.
A. Use Sensifast SYBR mastermix da Bioline (número de catálogo BIO-98050) para realizar PCR em cDNA preparado a partir de RNA de cérebro humano.
B. Use o instrumento CFX qPCR da Bio-Rad para registrar o mapa de amplificação e a curva de dissolução de apaleno (NM_033207, F: GCCATGGAGGAAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Gráfico de amplificação e curva de fusão de ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Gráfico de amplificação e curva de dissolução de GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs registrados em diferentes temperaturas de recozimento, mostrando a diferença em Cq registrada sob um gradiente de temperatura de 7C.
P 6 mostra um resultado típico de um teste indesejável, onde a qPCR foi realizada usando um gradiente Tas entre 59C e 67C (P 6a), usando primers para três genes específicos do cérebro humano.
Pode ser visto no gráfico de amplificação que os primers Opalin estão longe do ideal porque sua faixa Ta ideal é muito estreita (Figura 6b), ou seja, os Cqs são amplamente dispersos, resultando em Cqs sendo significativamente comparados aos seus Cqs baixos ideais.
Este método de detecção é instável e pode levar a uma amplificação abaixo do ideal.Portanto, este par de primers deve ser redesenhado.Além disso, a análise da curva de fusão (inserção) mostra que a especificidade desse método de detecção também pode ser problemática, porque a curva de fusão de cada Ta é diferente.
O método de detecção ACSBG1 mostrado em P 6c é mais robusto do que o método de detecção Opalin acima, mas ainda está longe do ideal e é provável que possa ser melhorado.
No entanto, enfatizamos que não há conexão necessária entre robustez e especificidade, pois a curva de dissolução produzida por este método de detecção mostra o mesmo valor de pico em todos os Tas (inserção).
Por outro lado, o teste de robustez é muito mais tolerante, produzindo Cqs semelhantes em uma ampla faixa de Tas, como no teste GFAP mostrado em P 6d.
A diferença em Cqs obtida na mesma faixa de 8 graus Celsius é menor que 1, e a curva de dissolução (inserção) confirma as características de detecção nessa faixa de temperatura.Vale a pena notar que a Tas calculada e a faixa de Ta real podem ser muito diferentes.
Existem muitas diretrizes projetadas para ajudar os pesquisadores a projetar primers eficientes, a maioria das quais é baseada em regras estabelecidas há muito tempo e muita atenção foi dada ao final 3' dos primers.Muitas vezes, é recomendado incluir um G ou C na extremidade 3' e duas bases G ou C (grampo GC), mas não mais do que duas das últimas 5 bases.
Na prática, essas regras podem orientar os pesquisadores, mas não são necessariamente corretas em todas as circunstâncias.
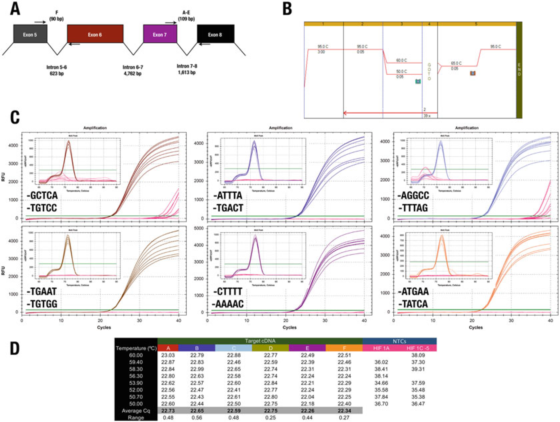
P7 |A extremidade 3' do primer tem pouco efeito na especificidade ou eficiência.
A. A posição dos primers para o gene humano HIF-1α (NM_181054.2).
B. Use o licor-mãe Agilent Brilliant III SYBR Green (Cat. No. 600882) para amplificar seis itens de teste.
C. Gráfico de amplificação e curva de fusão registrada pelo instrumento CFX qPCR da Bio-Rad e primers 3'end.NTCs são mostrados em vermelho.
D. Registro Cqs de cada item de teste
Por exemplo, o resultado em P 7 contradiz a regra 3'end.Todos os designs produzem basicamente os mesmos resultados, com apenas duas combinações de primers levando a uma amplificação não específica em NTC.
No entanto, não podemos apoiar o efeito do clipe GC, porque, neste caso, usar A ou T como máximo de 30 bases não reduz a especificidade.
O teste C, onde o primer F termina em GGCC, registrou Cqs em NTCs, indicando que se pode querer evitar essas sequências no final 30.Enfatizamos que a única maneira de determinar a melhor sequência 3'end de um par de primers é avaliar alguns primers candidatos experimentalmente.
Eficiência de amplificação
É importante ressaltar que, embora a detecção de PCR não específica nunca possa se tornar específica, a eficiência da amplificação pode ser ajustada e maximizada de várias maneiras diferentes, alterando a enzima, o licor mãe, os aditivos e as condições de ciclagem.
Para avaliar a eficiência da detecção por PCR, é melhor usar uma diluição seriada de 10 ou 5 vezes o ácido nucléico alvo, ou seja, o “método da curva padrão”.
Se amplicons de PCR ou alvos de DNA sintético forem usados para gerar uma curva padrão, diluições seriadas desses alvos devem ser misturadas com uma quantidade constante de DNA de base (como DNA genômico).
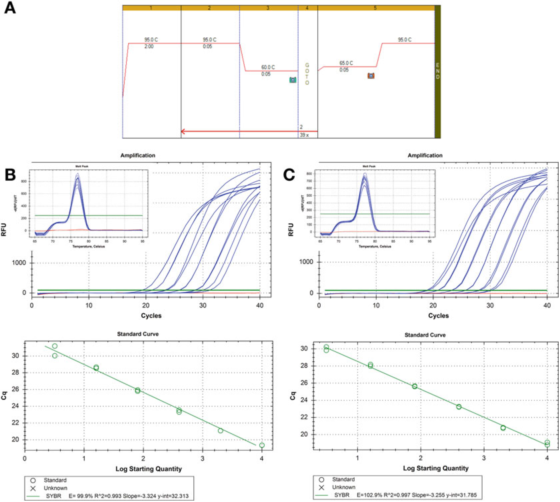
P8 |Curva de diluição para avaliar a eficiência da PCR.
A. Use primers para HIF-1: F: AAGAACTTTTAGGCCGCTCA e R: TGTCCTGTGGTGACTTGTCC e o mastermix Brilliant III SYBR Green da Agilent (número de catálogo 600882) para PCR e condições de curva de fusão.
B. 100 ng de RNA foram transcritos reversamente, diluídos 2 vezes, e amostras de cDNA diluídas em série foram diluídas 5 vezes para 1 ng de DNA genômico humano.A curva de fusão é mostrada na inserção.
C. A reação de RT, diluição e diluição em série foram repetidas para a segunda amostra de cDNA e os resultados foram semelhantes.
P 8 mostra duas curvas padrão, usando o mesmo método de detecção em duas amostras de cDNA diferentes, o resultado é a mesma eficiência, cerca de 100%, e o valor R2 também é semelhante, ou seja, o grau de ajuste entre os dados experimentais e a linha de regressão ou o grau de linearidade dos dados.
As duas curvas padrão são comparáveis, mas não exatamente iguais.Se o objetivo for quantificar com precisão o alvo, deve-se observar que é inaceitável fornecer um cálculo do número de cópias sem explicar a incerteza
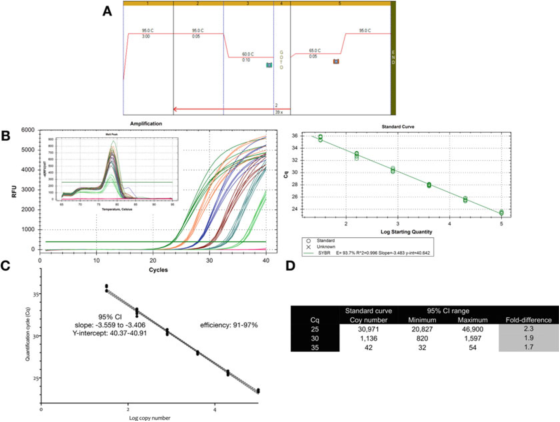
P9 |Incerteza de medição associada à quantificação usando uma curva padrão.
A. Use primers para GAPDH (NM_002046) para realizar PCR e condições de curva de fusão.F: ACAGTTGCCATGTAGACC e R: TAACTGGTTGAGCACAGG e Sensifast SYBR mastermix da Bioline (número de catálogo BIO-98050).
B. Gráfico de amplificação, curva de fusão e curva padrão registrada com o instrumento CFX qPCR da Bio-Rad.
C. Gráfico da curva padrão e intervalo de confiança (IC) de 95%.
D. O número de cópias e intervalo de confiança de 95% dos três valores Cq derivados da curva de diluição.
P 9 mostra que para um teste otimizado, a variabilidade inerente de uma única curva padrão é de aproximadamente 2 vezes (intervalo de confiança de 95%, mínimo a máximo), que pode ser a menor variabilidade que pode ser esperada.
Produto relacionado:
Kit qPCR de RT direto da célula
Kit de PCR direto de rabo de rato
Kit PCR direto de tecido animal
Horário da postagem: 30 de setembro de 2021