Em experimentos de qPCR, o design do primer também é um elo muito importante.Se os primers são adequados ou não, está intimamente relacionado a se a eficiência da amplificação atinge o padrão, se os produtos amplificados são específicos e se os resultados experimentais estão disponíveis.
Então, como melhorar a especificidade do primer qPCR?Alta eficiência de amplificação?
Hoje, levaremos você a projetar primers qPCR juntos e deixar que o design de primers qPCR se torne uma habilidade de conhecimento eficiente em experimentos.
Ao projetar os primers qPCR, geralmente preste atenção aos seguintes pontos: os primers devem ser projetados nos íntrons o máximo possível, o comprimento do produto deve ser de 100 a 300 pb, o valor de Tm deve ser o mais próximo possível de 60°C e os primers a montante e a jusante devem estar o mais próximo possível e o final do primer deve ser G ou C, etc. aguarde.
1. Projeto de primers abrangendo íntrons
Ao projetar primers qPCR, a escolha de primers projetados através de íntrons pode impedir que o modelo de gDNA seja amplificado, e os produtos são todos derivados da amplificação do cDNA, eliminando assim a influência da contaminação do gDNA.
2. Comprimento do iniciador
O comprimento do primer é geralmente entre 18-30 nt, e o comprimento do produto de amplificação deve ser controlado entre 100-300 bp tanto quanto possível.
Se o primer for muito curto, levará a uma amplificação inespecífica e, se for muito longo, formará facilmente uma estrutura secundária (como estrutura em grampo de cabelo).Se o produto de amplificação for muito longo, não é adequado para a reação da polimerase, o que afetará a eficiência da amplificação por PCR.
3. Conteúdo GC e valor Tm
O conteúdo de GC dos primers deve ser controlado entre 40% e 60%.Se for muito alto ou muito baixo, não é propício para iniciar a reação.O conteúdo de GC dos primers direto e reverso deve ser próximo ao mesmo para obter o mesmo valor de Tm e temperatura de recozimento.
O valor de Tm deve estar entre 55-65°C tanto quanto possível, geralmente em torno de 60°C, e o valor de Tm a montante e a jusante deve ser o mais próximo possível, de preferência não superior a 4°C.
4. Evite selecionar A na extremidade 3' do iniciador
Quando a extremidade 3' do primer é incompatível, há grandes diferenças na eficiência de síntese de diferentes bases.Quando a última base é A, ela também pode iniciar a síntese de cadeia mesmo no caso de incompatibilidade, e quando a última base é T Quando , a eficiência da indução de incompatibilidade é bastante reduzida.Portanto, evite escolher A na extremidade 3' do primer e é melhor escolher T.
Se for um primer de sonda, a extremidade 5' da sonda não pode ser G, porque mesmo quando uma única base G é conectada ao grupo repórter fluorescente FAM, G também pode extinguir o sinal fluorescente emitido pelo grupo FAM, resultando em resultados falsos negativos.Aparecer.
5. Distribuição base
A distribuição das quatro bases no primer é preferencialmente aleatória, evitando mais de 3 G ou C consecutivos na extremidade 3' e mais de 3 consecutivosG ou C são fáceis de gerar emparelhamento na região de sequência rica em GC.
6. A região do desenho do primer deve evitar estruturas secundárias complexas.
A estrutura secundária formada pela fita simples do produto de amplificação afetará o bom andamento da PCR.Prevendo antecipadamente se há uma estrutura secundária na sequência alvo, tente evitar essa região no desenho dos primers.
7. Os próprios primers e entre os primers devem tentar evitar bases complementares consecutivas.
Não pode haver complementaridade consecutiva de 4 bases entre o próprio primer e o primer.O primer em si não deve ter uma sequência complementar, caso contrário, ele se dobrará para formar uma estrutura em grampo, o que afetará a combinação de recozimento do primer e do molde.
Sequências complementares não podem existir entre primers upstream e downstream.A complementaridade entre primers produzirá dímeros de primer, o que reduzirá a eficiência da PCR e até afetará a precisão quantitativa.Se as estruturas de primer-dímero e hairpin forem inevitáveis, o valor △G não deve ser muito alto (deve ser inferior a 4,5 kcal/mol).
8. Os primers amplificam o produto específico alvo.
O objetivo final da detecção qPCR é entender a abundância do gene alvo.Se ocorrer amplificação não específica, a quantificação será imprecisa.Portanto, depois que os primers são projetados, eles precisam ser testados pelo BLAST, e a especificidade dos produtos é comparada no banco de dados de sequências.
Em seguida, tomamos o gene GAS6 humano (6 específico de parada de crescimento) como exemplo para projetar primers qPCR.
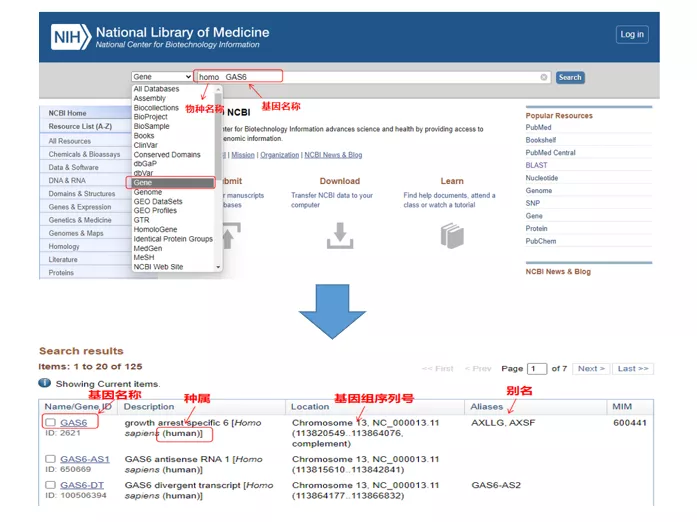
01 gene de consulta
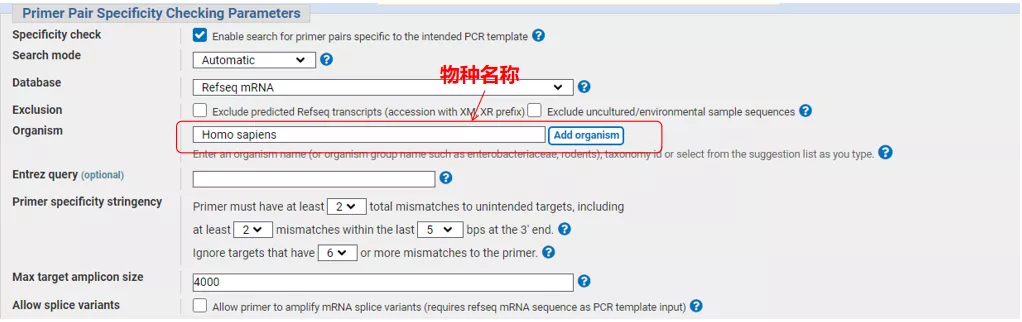
Homo GAS6através do NCBI.Aqui, devemos prestar atenção ao comparar o nome do gene e as espécies para garantir que sejam consistentes.
 02 Encontre a sequência do gene
02 Encontre a sequência do gene
(1) Se a sequência alvo for DNA genômico, selecione a primeira, que é a sequência de DNA genômico do gene.
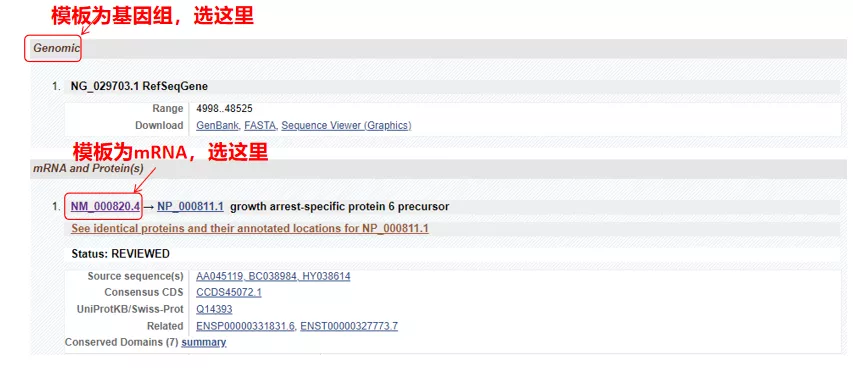 (2) Se a sequência alvo for mRNA, selecione a segunda.Após entrar, clique em “CDS” na tabela abaixo.A sequência de fundo marrom é a sequência de codificação do gene.
(2) Se a sequência alvo for mRNA, selecione a segunda.Após entrar, clique em “CDS” na tabela abaixo.A sequência de fundo marrom é a sequência de codificação do gene.
 03 Primários de design
03 Primários de design
Entre na interface do Primer-BLAST
 Digite o número da sequência do gene ou a sequência no formato Fasta no canto superior esquerdo e preencha os parâmetros relevantes.
Digite o número da sequência do gene ou a sequência no formato Fasta no canto superior esquerdo e preencha os parâmetros relevantes.
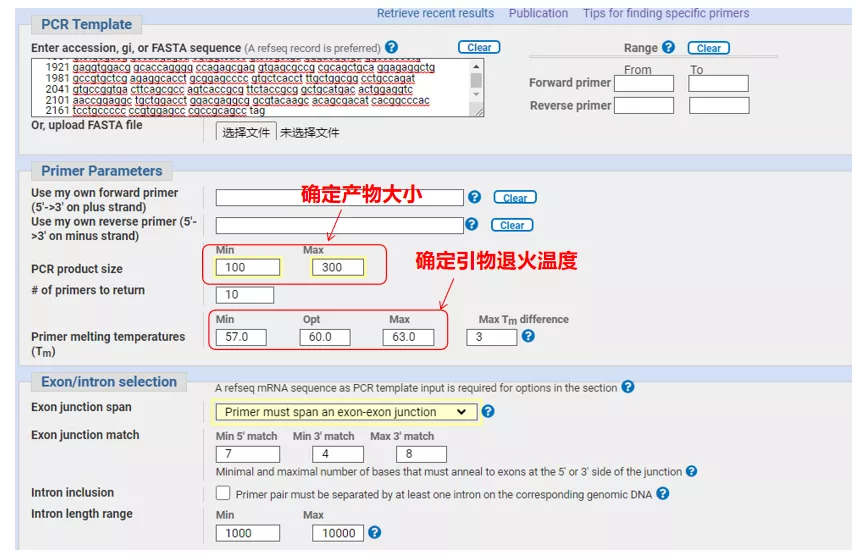
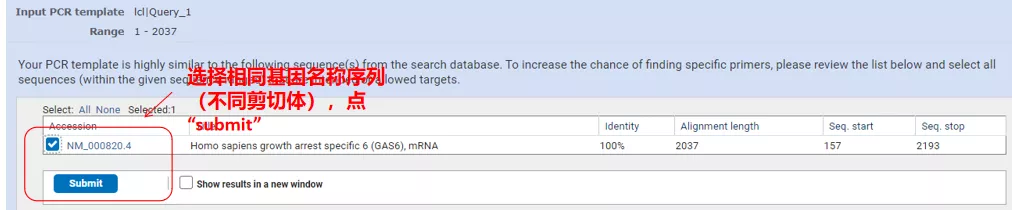
Clique em "Obter primers" e o NCBI aparecerá para informar que essa seleção de parâmetro será amplificada para outras variantes de emenda.Podemos verificar as diferentes variantes de splicing e enviá-las para obter o par de primers apropriado (conforme mostrado na figura abaixo).Esse processo pode levar dezenas de segundos para ser executado.
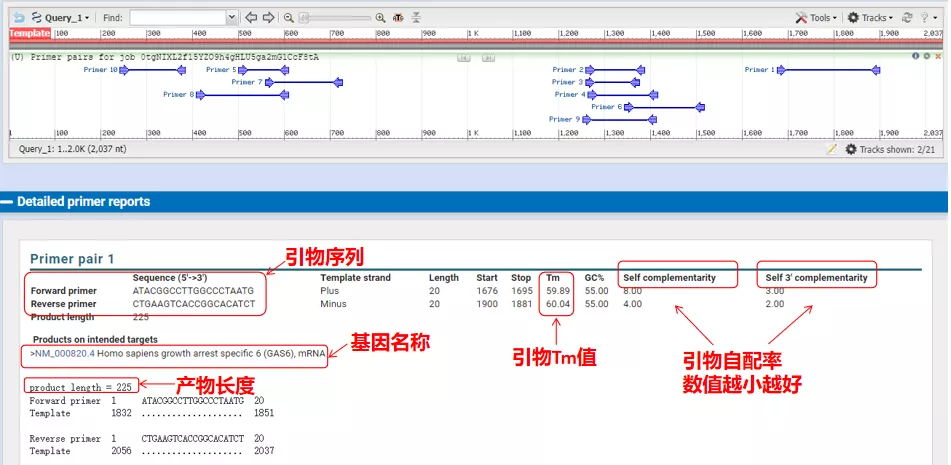 As temperaturas de recozimento desses pares de primers estão em torno de 60°C.De acordo com o objetivo do experimento, escolha primers com comprimento moderado, boa especificidade e menos autocomplementação dos primers para o experimento, e a taxa de sucesso é bastante alta!
As temperaturas de recozimento desses pares de primers estão em torno de 60°C.De acordo com o objetivo do experimento, escolha primers com comprimento moderado, boa especificidade e menos autocomplementação dos primers para o experimento, e a taxa de sucesso é bastante alta!
04Verificação da especificidade do primer
De fato, além de projetar primers, o Primer-Blast também pode avaliar os primers que nós mesmos projetamos.Retorne à página de design do primer, insira os primers upstream e downstream que projetamos e outros parâmetros não serão ajustados.Depois de enviar, você pode ver se o par de primers também existe em outros genes.Se todos eles forem exibidos no gene que queremos amplificar, isso indica que a especificidade desse par de primers é grande!(Por exemplo, este é o único resultado da consulta inicial!)
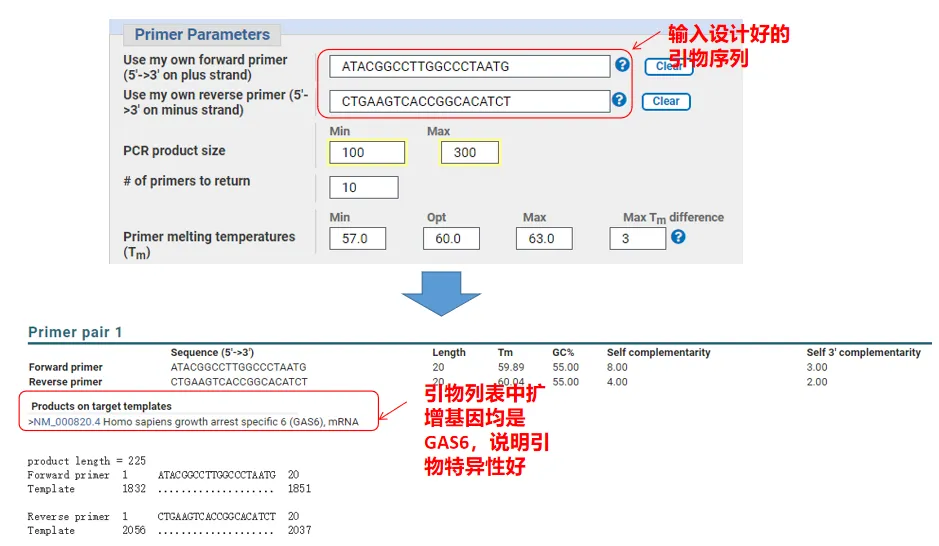
05 Julgamento de qualidade do primer
Que tipo de primer é o primer “perfeito” que combina “eficiência de amplificação até o padrão”, “características do produto amplificado” e “resultados experimentais confiáveis”?
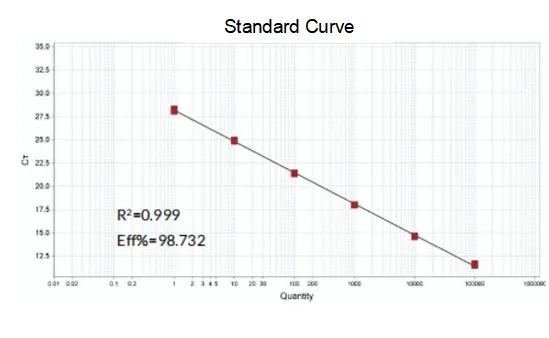 Eficiência de amplificação
Eficiência de amplificação
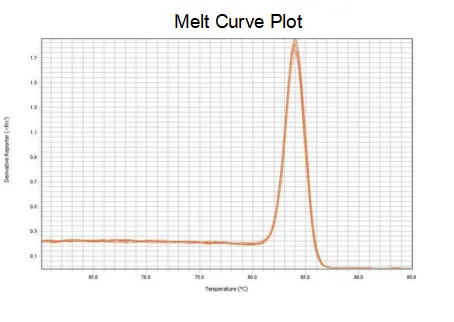 curva de fusão
curva de fusão
A eficiência de amplificação dos primers atinge 90%-110%, o que significa que a eficiência de amplificação é boa, e a curva de fusão tem um único pico e geralmente Tm>80°C, o que significa que a especificidade de amplificação é boa.
Produtos relacionados:
PCR em tempo real Easy–SYBR GREEN I
PCR em tempo real Easy-Taqman
Horário da postagem: 10 de fevereiro de 2023








