As três letras SNP são onipresentes no estudo da genética de populações.Independentemente da pesquisa de doenças humanas, posicionamento de características de culturas, evolução animal e ecologia molecular, os SNPs são necessários como base.No entanto, se você não tem uma compreensão profunda da genética moderna baseada em sequenciamento de alto rendimento, e diante dessas três letras, parece "o estranho mais familiar", então você não pode realizar a pesquisa subseqüente.Portanto, antes de embarcar na pesquisa de acompanhamento, vamos dar uma olhada no que é o SNP.
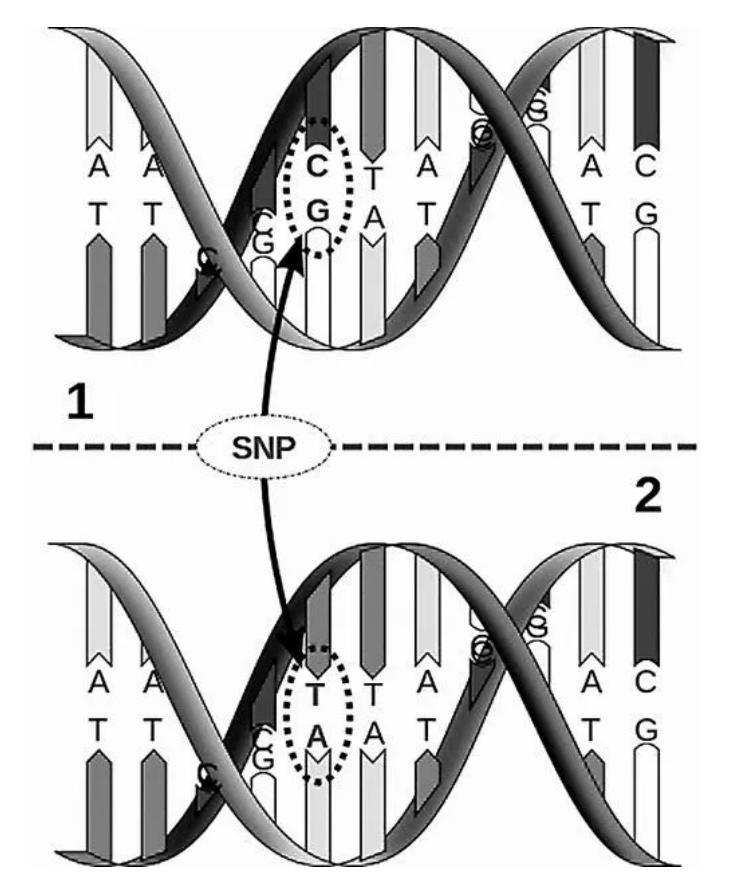
SNP (polimorfismo de nucleotídeo único), podemos ver em seu nome completo em inglês, refere-se à variação ou polimorfismo de nucleotídeo único.Ele também tem um nome separado, chamado SNV (variação de nucleotídeo único).Em alguns estudos humanos, apenas aqueles com frequência populacional superior a 1% são chamados de SNPs, mas, de maneira geral, os dois podem ser misturados.Assim, podemos dizer que o SNP, polimorfismo de nucleotídeo único, refere-se à mutação em que um nucleotídeo do genoma é substituído por outro nucleotídeo.Por exemplo, na figura abaixo, um par de bases AT é substituído por um par de bases GC, que é um sítio SNP.
Foto
No entanto, se é um "polimorfismo de nucleotídeo único" ou uma "variação de nucleotídeo único", é relativamente falando, então os dados SNP precisam do ressequenciamento do genoma como base, ou seja, os dados de sequenciamento são sequenciados após o sequenciamento do genoma do indivíduo.Comparado ao genoma, o sítio que difere do genoma é detectado como um sítio SNP.
Em termos de SNPs em vegetais,o kit Plant Direct PCRpode ser usado para detecção rápida.
Em termos de tipos de mutação, o SNP inclui transição e transversão.A transição refere-se à substituição de purinas por purinas ou pirimidinas por pirimidinas.A transversão refere-se à substituição mútua entre purinas e pirimidinas.A frequência de ocorrência será diferente e a probabilidade de transição será maior do que a de transversão.
Em termos de onde o SNP ocorre, diferentes SNPs terão efeitos diferentes no genoma.SNPs que ocorrem na região intergênica, ou seja, a região entre os genes no genoma, podem não afetar a função do genoma, e mutações no íntron ou na região promotora a montante do gene podem ter um certo impacto no gene;As mutações que ocorrem nas regiões de exon dos genes, dependendo se causam alterações nos aminoácidos codificados, têm efeitos diferentes nas funções dos genes.(Claro, mesmo que dois SNPs causem diferenças nos aminoácidos, eles têm efeitos diferentes na estrutura da proteína e, em última análise, os efeitos no fenótipo biológico podem ser bem diferentes).
No entanto, o número de SNPs que ocorrem em localizações de genes costuma ser significativamente menor do que em localizações não gênicas, pois um SNP que afeta a função de um gene costuma ter um impacto negativo na sobrevivência do indivíduo, fazendo com que o indivíduo portador desse SNP no grupo entre eles fosse eliminado.
Claro, para organismos diploides, os cromossomos existem em pares, mas é impossível que um par de cromossomos seja exatamente o mesmo para todas as bases.Portanto, alguns SNPs também aparecerão como heterozigotos, ou seja, há duas bases nessa posição do cromossomo.Em um grupo, os genótipos SNP de diferentes indivíduos são agregados, o que se torna a base para a maioria das análises subsequentes.Em combinação com as características, pode-se julgar se o SNP como um marcador molecular está ligado às características, o QTL (locus quantitativo da característica) da característica pode ser julgado e GWAS (estudo de associação do genoma) ou a construção do mapa genético pode ser realizada;SNP pode ser usado como marcador molecular Julgar a relação evolutiva entre indivíduos;você pode rastrear SNPs funcionais e estudar mutações relacionadas a doenças;você pode usar mudanças de frequência de alelos SNP ou taxas heterozigóticas e outros indicadores para determinar as regiões selecionadas no genoma... etc., combinadas com o presente Com o desenvolvimento do sequenciamento de alto rendimento, centenas de milhares ou mais locais SNP podem ser obtidos de um conjunto de dados de sequenciamento.Pode-se dizer que o SNP agora se tornou a pedra angular da pesquisa em genética populacional.
Claro, as mudanças nas bases do genoma nem sempre são a substituição de uma base por outra (embora esta seja a mais comum).Também é possível que faltem uma ou algumas bases, ou duas bases.Várias outras bases foram inseridas no meio.Essa pequena faixa de inserções e deleções é chamada coletivamente de InDel (inserção e deleção), que se refere especificamente à inserção e deleção de fragmentos curtos (uma ou várias bases).O InDel que ocorre no local de um gene também pode ter um impacto na função do gene; portanto, às vezes, o InDel também pode desempenhar um papel importante na pesquisa.Mas, no geral, o status do SNP como pedra angular da genética populacional ainda é inabalável.
Horário da postagem: 27 de agosto de 2021