A vacina de mRNA da Pfizer para COVID reacendeu a paixão pelo uso do ácido ribonucleico (RNA) como alvo terapêutico.No entanto, direcionar o RNA com moléculas pequenas é extremamente desafiador.
O RNA possui apenas quatro blocos de construção: adenina (A), citosina (C), guanina (G) e uracila (U), que substitui a timina (T) encontrada no DNA.Isso torna a seletividade dos medicamentos um obstáculo quase intransponível.Em contraste, existem 22 aminoácidos naturais que compõem as proteínas, o que explica por que a maioria dos medicamentos direcionados a proteínas tem uma seletividade relativamente boa.
Estrutura e função do RNA
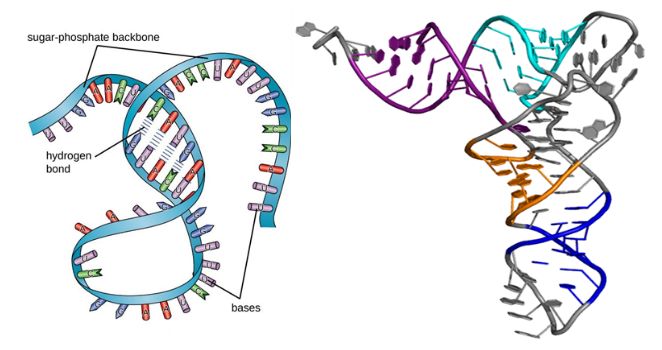
Assim como as proteínas, as moléculas de RNA possuem estruturas secundárias e terciárias, conforme mostrado na figura abaixo.Embora sejam macromoléculas de cadeia simples, sua estrutura secundária toma forma quando o pareamento de bases causa protuberâncias, loops e hélices.Então, o dobramento tridimensional leva à estrutura terciária do RNA, que é essencial para sua estabilidade e função.
Figura 1. Estrutura do RNA
Existem três tipos de RNA:
- RNA mensageiro (mRNA)transcreve a informação genética do DNA e é transferida como uma sequência de bases para o ribossomo;eu
- RNA ribossomal (rRNA)faz parte das organelas sintetizadoras de proteínas chamadas ribossomos, que são exportadas para o citoplasma e ajudam a traduzir as informações do mRNA em proteínas;
- RNA de transferência (tRNA)é a ligação entre o mRNA e a cadeia de aminoácidos que compõe a proteína.
Direcionar o RNA como um alvo terapêutico é muito atraente.Verificou-se que apenas 1,5% do nosso genoma é traduzido em proteína, enquanto 70% a 90% é transcrito em RNA.As moléculas de RNA são as mais importantes para todos os organismos vivos.De acordo com o “dogma central” de Francis Crick, o papel mais crítico do RNA é traduzir a informação genética do DNA em proteínas.Além disso, as moléculas de RNA também têm outras funções, incluindo:
- Atuando como moléculas adaptadoras na síntese de proteínas;eu
- Atuando como mensageiro entre o DNA e o ribossomo;eu
- Eles são portadores de informação genética em todas as células vivas;eu
- Promovendo a seleção ribossômica dos aminoácidos corretos, necessários para a síntese de novas proteínasna Vivo.
antibióticos
Apesar de ter sido descoberto já na década de 1940, o mecanismo de ação de muitos antibióticos não foi elucidado até o final da década de 1980.Verificou-se que uma grande proporção de antibióticos age ligando-se aos ribossomos bacterianos para impedi-los de produzir proteínas apropriadas, matando assim as bactérias.
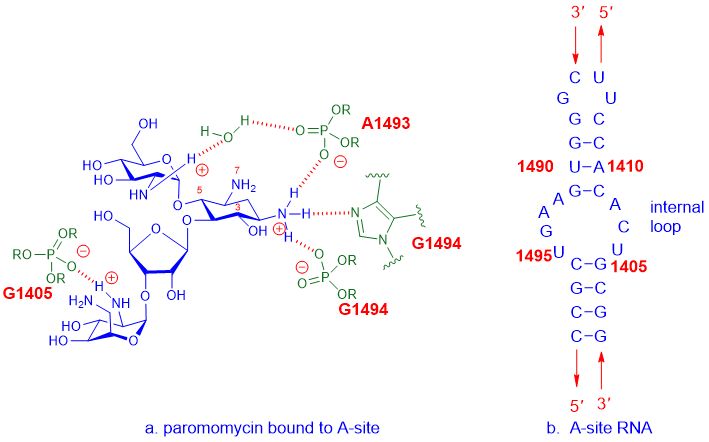
Por exemplo, os antibióticos aminoglicosídeos se ligam ao sítio A do rRNA 16S, que faz parte da subunidade 30S do ribossomo, e então interferem na síntese de proteínas para interferir no crescimento bacteriano, levando à morte celular.O sítio A refere-se ao sítio aminoacil, também conhecido como sítio aceitador de tRNA.A interação detalhada entre medicamentos aminoglicosídeos, comoparomomicina, e o sítio A deE. coliO RNA é mostrado abaixo.
Figura 2. A interação entre a paromomicina e o sítio A deE. coliARN
Infelizmente, muitos inibidores do sítio A, incluindo medicamentos aminoglicosídeos, apresentam problemas de segurança, como nefrotoxicidade, dose-dependente e ototoxicidade irreversível específica.Essas toxicidades são o resultado de uma falta de seletividade em drogas aminoglicosídeos para reconhecer pequenas moléculas de RNA.
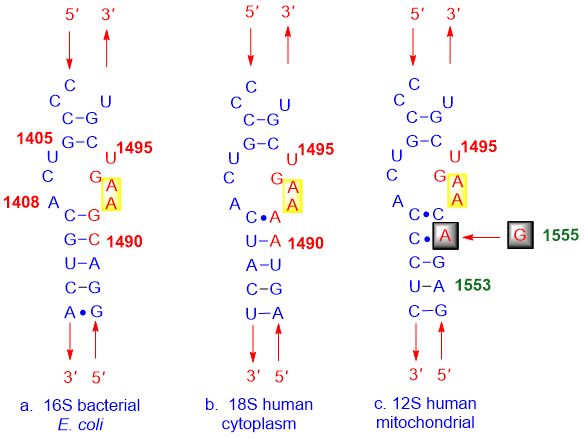
Conforme mostrado na figura abaixo: (a) a estrutura da bactéria, (b) a membrana celular humana e (c) o sítio A mitocondrial humano são muito semelhantes, fazendo com que os inibidores do sítio A se liguem a todos eles.
Figura 3. A ligação não seletiva do inibidor do sítio A
Os antibióticos de tetraciclina também inibem o sítio A do rRNA.Eles inibem seletivamente a síntese de proteínas bacterianas por ligação reversível a uma região helicoidal (H34) na subunidade 30S complexada com Mg2+.
Por outro lado, os antibióticos macrólidos se ligam perto do local de saída (E-site) do túnel do ribossomo bacteriano para peptídeos nascentes (NPET) e o bloqueiam parcialmente, inibindo assim a síntese de proteínas bacterianas.Finalmente, os antibióticos oxazolidinona, comolinezolida(Zyvox) se ligam a uma fenda profunda na subunidade ribossômica 50S bacteriana, que é cercada por nucleotídeos 23S rRNA.
Oligonucleotídeos antisense (ASO)
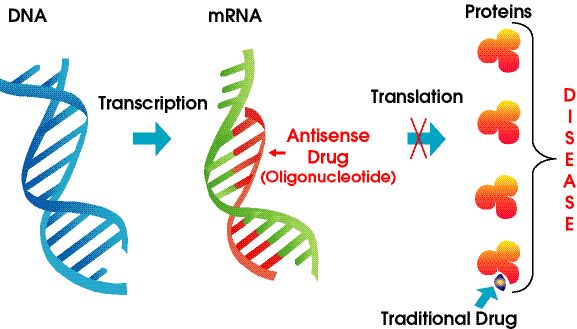
As drogas antisense são polímeros de ácidos nucleicos modificados quimicamente que têm como alvo o RNA.Eles dependem do pareamento de bases Watson-Crick para se ligar ao mRNA alvo, resultando em silenciamento de gene, bloqueio estérico ou alteração de splicing.ASOs podem interagir com pré-RNAs no núcleo da célula e mRNAs maduros no citoplasma.Eles podem ter como alvo éxons, íntrons e regiões não traduzidas (UTRs).Até o momento, mais de uma dúzia de medicamentos ASO foram aprovados pelo FDA.
Figura 4. Tecnologia Antisense
Medicamentos de moléculas pequenas direcionados ao RNA
Em 2015, a Novartis relatou ter descoberto um regulador de splicing SMN2 chamado Branaplam, que aumenta a associação de U1-pré-mRNA e resgata camundongos SMA.
Por outro lado, o Risdiplam (Evrysdi) da PTC/Roche foi aprovado pelo FDA em 2020 para o tratamento da SMA.Como o Branaplam, o Risdiplam também funciona regulando o splicing de genes SMN2 relevantes para produzir proteínas SMN funcionais.
degradadores de RNA
RBM significa RNA-binding motif protein.Essencialmente, a indol sulfonamida é um adesivo molecular.Recruta seletivamente o RBM39 para a ubiquitina ligase CRL4-DCAF15 E3, promovendo a poliubiquitinação do RBM39 e a degradação da proteína.A depleção genética ou a degradação mediada por sulfonamidas de RBM39 induz anormalidades significativas de splicing em todo o genoma, levando, em última análise, à morte celular.
Os RNA-PROTACs são desenvolvidos para degradar as proteínas de ligação ao RNA (RBPs).O PROTAC usa um ligante para conectar o ligante da ligase E3 ao ligante do RNA, que se liga ao RNA e às RBPs.Como o RBP contém domínios estruturais que podem se ligar a sequências oligonucleotídicas específicas, o RNA-PROTAC usa uma sequência oligonucleotídica como ligante para a proteína de interesse (POI).O resultado final é a degradação dos RBPs.
Recentemente, o professor Matthew Disney, da Scripps Institution of Oceanography, inventou o RNAquimeras de direcionamento de ribonuclease (RiboTACs).RiboTAC é uma molécula heterofuncional que conecta um ligante de RNase L e um ligante de RNA com um ligante.Ele pode recrutar especificamente RNase L endógena para alvos de RNA específicos e, em seguida, eliminar com sucesso o RNA usando o mecanismo de quebra de ácido nucleico celular (RNase L).
À medida que os pesquisadores aprendem mais sobre a interação entre pequenas moléculas e alvos de RNA, mais drogas usando esse método surgirão no futuro.
Horário da postagem: 02 de agosto de 2023