Depois que o estudioso americano Eric S. Lander propôs formalmente o polimorfismo de nucleotídeo único (SNP) como o marcador molecular de terceira geração em 1996, o SNP tem sido amplamente utilizado na análise de associação de traços econômicos, construção de mapas de ligação genética biológica e triagem de genes patogênicos humanos., Diagnóstico e previsão de risco de doenças, triagem individualizada de medicamentos e outros campos de pesquisa biológica e médica.No campo de cultivo comercial, a detecção de SNP pode realizar a seleção precoce de características necessárias.Esta seleção tem as características de alta precisão e pode efetivamente evitar a interferência da morfologia e fatores ambientais, encurtando assim o processo de reprodução.Portanto, SNP desempenha um papel enorme no campo da pesquisa básica.
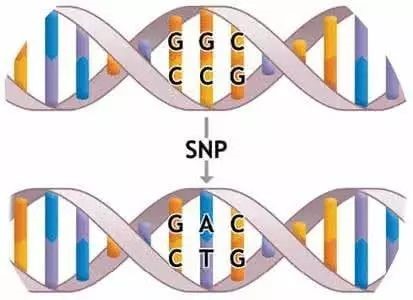
Polimorfismo de nucleotídeo único (Single Nucleotide Polymorphism, SNP) refere-se ao fenômeno de que existem diferenças de nucleotídeo único na mesma posição na sequência de DNA de indivíduos da mesma espécie ou de espécies diferentes.A inserção, exclusão, conversão e inversão de uma única base podem causar essa diferença.No passado, a definição de SNP era diferente da de mutação.Um locus variante requer que a frequência de um dos alelos na população seja maior que 1% para ser definido como um locus SNP.No entanto, com a expansão das teorias biológicas modernas e a aplicação da tecnologia, a frequência alélica não é mais uma condição necessária para limitar a definição de SNP.De acordo com os dados de variação de nucleotídeo único incluídos no banco de dados de polimorfismos de nucleotídeo único (dbSNP) do National Center for Biotechnology Information (NCBI), inserção/exclusão de baixa frequência, variação de microssatélite, etc. também estão incluídos.
No corpo humano, a frequência de SNP é de 0,1%.Em outras palavras, há uma média de um site SNP por 1000 pares de bases.Embora a frequência de ocorrência seja relativamente alta, nem todos os sítios SNP podem ser candidatos a marcadores relacionados a características.Isso está relacionado principalmente ao local onde o SNP ocorre.
Teoricamente, o SNP pode ocorrer em qualquer lugar na sequência do genoma.Os SNPs que ocorrem na região codificadora podem produzir mutações sinônimas e mutações não sinônimas, ou seja, o aminoácido muda ou não muda antes e depois da mutação.O aminoácido alterado geralmente faz com que a cadeia peptídica perca sua função original (mutação sem sentido) e também pode interromper a tradução (mutação sem sentido).Os SNPs que ocorrem em regiões não codificantes e regiões intergênicas podem afetar o splicing do mRNA, a composição da sequência do RNA não codificante e a eficiência de ligação dos fatores de transcrição e do DNA.A relação específica é mostrada na figura:
Tipos de SNP:
Vários métodos comuns de tipagem SNP e sua comparação
De acordo com diferentes princípios, os métodos comuns de detecção de SNP são divididos nas seguintes categorias:
Comparação de classificação de métodos de detecção
Nota: Listados na tabela estão os métodos de detecção de SNP mais comuns atualmente usados, outros métodos de detecção, como hibridação de local específico (ASH), extensão de iniciador de local específico (ASPE), extensão de base única (SBCE), corte de local específico (ASC), tecnologia de chip de gene, tecnologia de espectrometria de massa, etc. não foram classificados e comparados.
O custo e o tempo de purificação do ácido nucleico nos vários métodos comuns de detecção de SNP acima são inevitáveis.No entanto, os kits relacionados baseados na tecnologia de PCR direta da Foregene podem realizar diretamente a amplificação por PCR ou qPCR em amostras não purificadas, o que traz uma conveniência sem precedentes para a detecção de SNP.
Os produtos da série de PCR direto da Foregene omitem simples e grosseiramente as etapas de purificação da amostra, o que reduz muito o tempo e o custo necessários para preparar os modelos.A exclusiva Taq polimerase possui excelente capacidade de amplificação e pode tolerar uma variedade de inibidores de ambientes de amplificação complexos.Estas características fornecem uma garantia técnica para a obtenção de produtos específicos de alto rendimento.Foregene Direct PCR/qPCR kits para vários tipos de amostras, tais como: tecidos animais (rabo de rato, zebrafish, etc.), folhas de plantas, sementes (incluindo amostras de polissacarídeos e polifenóis), etc.
Horário da postagem: 23 de julho de 2021