Como novo no laboratório, não é um bom trabalho separar plantas positivas de um grupo de plantas com baixa taxa de conversão.Em primeiro lugar, o DNA deve ser extraído de um grande número de amostras uma a uma e, em seguida, os genes estranhos serão detectados por PCR.No entanto, os resultados geralmente são espaços em branco e faixas com alguns itens ocasionalmente, mas é impossível determinar se há detecções perdidas ou falsas..É muito impotente enfrentar tal processo experimental e resultados?Não se preocupe, o irmão ensina como filtrar plantas transgênicas positivas com facilidade e precisão.
Passo 1
Iniciadores de detecção de design
Determine o gene endógeno e o gene exógeno a serem detectados de acordo com a amostra a ser testada e selecione uma sequência representativa de 100-500 pb no gene para o design do primer.Bons primers podem garantir a precisão dos resultados da detecção e encurtar o tempo de detecção (consulte o apêndice para os primers de detecção comumente usados).
Aviso: Os primers recém-projetados precisam otimizar as condições de reação e verificar a exatidão, precisão e limite de detecção da detecção antes da detecção em grande escala.
Passo 2
Projetar protocolo experimental
Controle positivo: Use o DNA purificado contendo o fragmento alvo como modelo para determinar se o sistema de reação de PCR e as condições são normais.
Controle negativo/branco: Use o molde de DNA ou ddH2O que não contém o fragmento alvo como molde para detectar se há uma fonte de contaminação no sistema de PCR.
Controle interno de referência: use a combinação primer/sonda do gene endógeno da amostra a ser testada para avaliar se o molde pode ser detectado por PCR.
Perceber:
Controles positivos, negativos/em branco e controles de controle interno devem ser definidos para cada teste para avaliar a validade dos resultados experimentais.
Preparação do experimento
Antes de usar, observe se a solução está uniformemente misturada.Se for encontrada precipitação, ela precisa ser dissolvida e misturada de acordo com as instruções antes do uso.A mistura 2×PCR precisa ser pipetada e misturada repetidamente com uma micropipeta antes do uso para evitar distribuição desigual de íons.
Perceber:
Pegue o manual e leia-o cuidadosamente, e faça os preparativos antes do experimento em estrita conformidade com os requisitos do manual.
Passo 4
Preparar o sistema de reação de PCR
De acordo com o protocolo experimental, misture os primers, H2O e 2×PCR misture uniformemente, centrifugue e distribua-os para cada tubo de reação.
Perceber:
Para testes em larga escala ou de longo prazo, recomenda-se usar um sistema de reação de PCR contendo enzima UNG, que pode efetivamente evitar a contaminação por aerossol causada por produtos de PCR.
Passo 5
Adicionar modelo de reação
Usando a tecnologia Direct PCR, não há necessidade de tedioso processo de purificação de ácido nucleico, o modelo de amostra pode ser preparado em 10 minutos e o sistema de reação de PCR correspondente pode ser adicionado.
Perceber:
O método de clivagem tem melhor efeito de detecção e o produto obtido pode ser usado para múltiplas reações de detecção.
5.1: Expansão direta das folhas
De acordo com o tamanho da imagem no manual, corte o tecido da folha com um diâmetro de 2-3mm e coloque-o no sistema de reação de PCR.
Observação: Certifique-se de que os fragmentos de folha estejam completamente imersos na solução de reação de PCR e não adicione tecido foliar em excesso.
5.2: Método de divisão da folha
Corte o tecido foliar com um diâmetro de 5-7mm e coloque-o em um tubo de centrífuga.Se você escolher folhas maduras, evite usar os tecidos da nervura principal da folha.Pipete 50ul de lisado de tampão P1 em um tubo de centrífuga para garantir que o lisado possa imergir completamente o tecido foliar, coloque-o em um termociclador ou banho de metal e lise a 95°C por 5 a 10 minutos.
Adicionar 50ul de solução de neutralização Buffer P2 e misturar bem.O lisado resultante pode ser usado como modelo e adicionado ao sistema de reação de PCR.
Nota: A quantidade de modelo está entre 5-10% do sistema de PCR e não deve exceder 20% (por exemplo, em um sistema de PCR de 20μl, adicione 1-2μl de solução de lise, não mais que 4μl).
Passo 6
reação de PCR
Depois de centrifugar o tubo de reação de PCR, ele é colocado em um instrumento de PCR para amplificação.
Perceber:
A reação usa molde não purificado para amplificação, então o número de ciclos de amplificação é de 5 a 10 ciclos a mais do que quando se usa molde de DNA purificado.
Passo 7
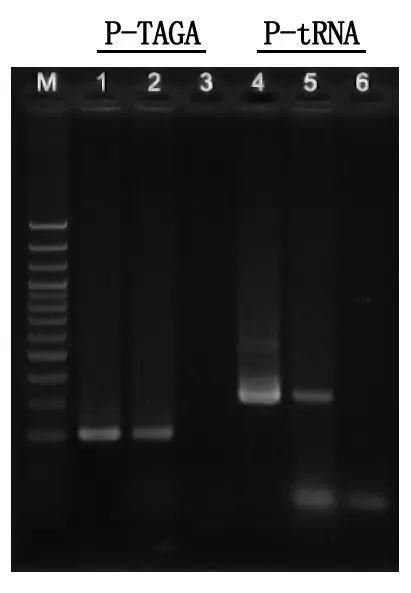
Detecção de eletroforese e análise de resultados
M: 100bp DNA Ladder
1\4: Método de DNA purificado
2\5: Método de PCR direto
3\6: controle em branco
controle de qualidade:
Os resultados do teste dos vários controles definidos no experimento devem atender às seguintes condições.Caso contrário, a causa do problema deve ser analisada e o teste deve ser realizado novamente após a eliminação do problema.
Tabela 1. Resultados de testes normais de vários grupos de controle
*Quando o plasmídeo é usado como controle positivo, o resultado do teste de gene endógeno pode ser negativo
Julgamento do resultado:
A. O resultado do teste do gene endógeno da amostra é negativo, indicando que o DNA adequado para detecção de PCR comum não pode ser extraído da amostra ou o DNA extraído contém inibidores da reação de PCR, e o DNA deve ser extraído novamente.
B. O resultado do teste do gene endógeno da amostra é positivo e o resultado do teste do gene exógeno é negativo, indicando que o DNA adequado para detecção de PCR comum foi extraído da amostra e pode-se julgar que o gene XXX não foi detectado na amostra.
C. O resultado do teste do gene endógeno da amostra é positivo e o resultado do teste do gene exógeno é positivo, indicando que o DNA adequado para detecção de PCR comum foi extraído da amostra e o DNA da amostra contém o gene XXX.Experimentos de confirmação podem ser realizados posteriormente.
Passo 8
Iniciadores de detecção de design
Após o experimento, use solução de hipoclorito de sódio a 2% e solução de etanol a 70% para limpar a área experimental para evitar a poluição ambiental。
Horário da postagem: 08 de setembro de 2021