PCR, múltiplo PCR, PCR in situ, PCR reverso, RT-PCR, qPCR(1)–PCR
Vamos resolver os conceitos, etapas e detalhes de vários PCR
Ⅰ. PCR
A Reação em Cadeia da Polimerase, conhecida como PCR, é uma tecnologia de biologia molecular usada para ampliar fragmentos de DNA específicos.Pode ser considerado como uma replicação especial do DNA in vitro.A DNA polimerase (DNA Polimerase I) foi descoberta já em 1955, e o Fragmento Klenow de E. Coli, que tem valor experimental e praticidade, foi descoberto pelo Dr.As enzimas em uso hoje (chamadas Taq polimerase), foram isoladas de Thermus aquaticus, uma bactéria de águas termais em 1976. Sua característica é resistir a altas temperaturas e é uma enzima ideal, mas é amplamente utilizada após os anos 1980.O conceito original do protótipo primitivo original de PCR é semelhante ao reparo e cópia de genes, que foi proposto pelo Dr. KJell Kleppe em 1971. Ele publicou a primeira cópia simples e de curto prazo do gene (semelhante aos dois primeiros ciclos de reações de PCR).O PCR desenvolvido hoje foi desenvolvido pelo Dr. Kary B. Mullis em 1983. O Dr. Mullis atendeu empresas de PE naquele ano, então o PE tem um status especial na indústria de PCR.O Dr. Mullis publicou oficialmente o primeiro artigo relacionado com Saiki e outros em 1985. Desde então, o uso de PCR é feito a milhares de quilômetros por dia, e pode-se dizer que a qualidade dos artigos relacionados torna muitos outros métodos de pesquisa intragáveis.Posteriormente, a tecnologia de PCR é amplamente utilizada em pesquisas científicas biológicas e aplicações clínicas, tornando-se a tecnologia mais importante da pesquisa em biologia molecular.Mullis também ganhou o Prêmio Nobel de Química de 1993.
PCRPrincípio
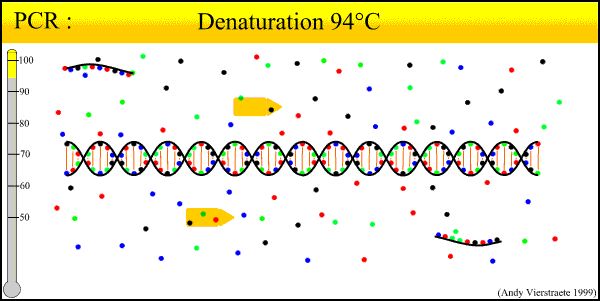
O princípio básico da tecnologia de PCR é semelhante ao processo de replicação natural do DNA, e sua especificidade depende do oligonucleotídeo iniciador que é complementar a ambas as extremidades da sequência alvo.A PCR é composta por três etapas básicas de reação de degeneração-recozimento-extensão: ①Degeneração do DNA modelo: Depois que o DNA modelo é aquecido a cerca de 93°C por um determinado período de tempo, a solução de DNA duplo para o DNA de cadeia dupla formada pela amplificação de PCR do DNA modelo Saindo, torne-o uma única cadeia para que possa ser combinado com o primer para se preparar para a próxima rodada de reação.②O recozimento (composto) do DNA modelo e do primer: Depois que o DNA modelo é aquecido e degenerado em uma única cadeia, a temperatura cai para cerca de 55°C.A sequência complementar do primer e do molde de DNA de cadeia simples.③A extensão do primer: modelo de DNA - a ligação do primer é baseada na ação da TaqDNA polimerase, com dNTP como matéria-prima da reação.Mantenha o princípio de replicação, sintetize uma nova cadeia de cópias semi-reservada que complemente a cadeia de DNA modelo e repita o ciclo degeneração-recozimento-extensão três processos podem obter mais “cadeia de cópias semi-reservadas” e esta nova cadeia está disponível novamente Torne-se um modelo para o próximo ciclo.Leva de 2 a 4 minutos para completar o loop, o gene alvo pode ser amplificado vários milhões de vezes em 2 a 3 horas.
PadrãoPCRSistema de Reação
| Taq DNA Polimerase | 2,5 μl |
| Mg2+ | 1,5mmol/L |
| 10 × buffer de amplificação | 10μl |
| 4 misturas dNTP | 200μl |
| DNA modelo | 0,1~2μg |
| cartilha | 10~100μl |
| Adicione água fervente dupla ou tripla | 100 μl |
Cinco elementos da reação de PCR
Existem principalmente cinco tipos de substâncias envolvidas na reação de PCR, ou seja, primer, enzima, dNTP, molde e tampão (Mg2+ é necessário).[procedimento de PCR]
O processo de PCR padrão é dividido em três etapas
1. Degeneração do DNA (90°C-96°C): Moldes de DNA de cadeia dupla sob ação térmica, as pontes de hidrogênio se quebram, formando um DNA de cadeia simples.
2. Recozimento (25℃ -65℃): A temperatura do sistema é reduzida, o primer é combinado com o modelo de DNA para formar uma cadeia dupla local.
3. Extensão (70℃ -75℃): Sob a ação da enzima Taq (cerca de 72°C, a melhor atividade), o dNTP é usado como matéria-prima, estende-se da extremidade 5' do iniciador → extremidade 3', a síntese e o modelo complementam-se na cadeia de DNA.
Cada ciclo é desnaturado, recozido e estendido, dobrando o conteúdo de DNA.Atualmente, devido à pequena área de amplificação, algumas PCR podem ser replicadas em um tempo muito curto, mesmo que a atividade da enzima Taq não seja ideal, portanto, pode ser alterada para duas etapas, ou seja, o recozimento e a extensão podem ser realizados a 60°C-65°C ao mesmo tempo.A fim de reduzir o processo de levantamento e resfriamento e melhorar a velocidade de resposta.
Recursos de reação de PCR
● Alta especificidade
Os fatores decisivos específicos da resposta de PCR são: ①A combinação específica do primer e do DNA molde.②O princípio do emparelhamento de bases.③A lealdade da reação de síntese da TaqDNA polimerase.④A especificidade e conservadorismo do gene alvo.
A combinação correta de primers e modelos é a chave.A ligação do primer e do molde e a extensão da cadeia do primer são baseadas no princípio da combinação de bases alcalinas.A fidelidade das reações de síntese da polimerase e a resistência a altas temperaturas da Taq DNA polimerase para fazer a ligação (composto) do molde e do primer na reação podem ser realizadas em uma temperatura mais alta.A especificidade da combinação é grandemente aumentada.O clipe pode manter um alto grau de correção.Ao selecionar uma região genética alvo com alta conservatividade e alta conservatividade, sua especificidade é maior.
● Alta sensibilidade
O volume de produção de produtos de PCR é aumentado pelo índice, que pode expandir o modelo inicial do Picker (PG=10-12) para aumentar o nível do microcontrolador para o nível de microgramas (μg= -6).Uma célula-alvo pode ser detectada a partir de 1 milhão de células;na detecção de vírus, a sensibilidade do PCR pode chegar a 3 RFUs (unidades formadas por manchas vazias);a taxa mínima de detecção na ciência bacteriana é de 3 bactérias.
● Simples e rápido
A reflexão de PCR usa uma Taq DNA polimerase de alta temperatura, que adiciona a solução de reação de uma só vez, ou seja, uma reação de degeneração-recozimento-extensão na solução de amplificação de DNA e pote de banho-maria.Geralmente, a reação de amplificação é concluída em 2 a 4 horas.Os produtos aumentados são geralmente analisados por espada elétrica e não precisam usar isótopos, sem poluição radioativa e fácil promoção.
● A pureza da amostra é baixa
Não há necessidade de separar vírus ou bactérias e células de cultura.Produtos brutos de DNA e RNA podem ser usados como amplificadores.A detecção de amplificação de DNA pode ser usada diretamente usando amostras clínicas, como sangue, líquido corporal, líquido para tosse, cabelo, células e tecidos vivos.
PCRproblemas comuns
● Falso negativo, sem bandas amplificadas
As principais etapas da reação de PCR incluem: ① preparação de ácidos nucleicos modelo, ② qualidade e especificidade dos primers, ③ qualidade das enzimas ④ condições do ciclo de PCR.Encontrar o motivo também deve ser analisado e estudado pelos links acima.
Modelos: ① O modelo contém diversas proteínas, ② O modelo contém um inibidor da enzima Taq, ③ A proteína no modelo não é eliminada, especialmente a proteína de grupo no cromossomo.⑤ A degeneração do ácido nucleico do Deminer não é completa.Quando a qualidade das enzimas e primers é boa, não há banda de amplificação, o que provavelmente é o tratamento digestivo das amostras.Há algo de errado com o processo de extração de ácido nucleico modelo, portanto, para preparar uma solução de digestão eficaz e estável, seu procedimento deve ser corrigido e não alterado arbitrariamente.
Inativação enzimática: uma nova enzima ou ambas antigas e novas enzimas devem ser usadas para analisar se a atividade enzimática é perdida ou insuficiente, levando a falsos negativos.Deve-se notar que a enzima Taq ou o brometo de etídio às vezes são esquecidos.
Primer: a qualidade do primer, a concentração do primer e se a concentração dos dois primers é simétrica.É um motivo comum para a falha do PCR ou a banda crescente não ser ideal e propensa a se difundir.Existem problemas com a qualidade dos primers de alguns números de lote.Os dois primers têm uma concentração alta e uma concentração baixa, causando amplificação assimétrica de baixa eficiência.As contramedidas são: ① Selecione um bom primer para sintetizar unidades.② A concentração do primer não depende apenas do valor OD, mas também presta atenção ao líquido original do primer para fazer a eletroforese em gel de ágar-açúcar.Deve haver uma zona de tira de primer e o brilho dos dois primers deve ser geralmente consistente.Belt, a PCR pode falhar neste momento e deve ser resolvida com a unidade de síntese do primer.Se um primer for alto, o brilho é baixo e sua concentração deve ser equilibrada quando diluída.③ O primer deve ser pago e armazenado em alta concentração para evitar congelamento múltiplo ou peças de refrigeração de longo prazo do refrigerador, o que fará com que o primer se deteriore e se degrade.④ O design do primer não é razoável, como o comprimento do primer é insuficiente e o agrupamento di é formado entre os primers.
Concentração de Mg2+: A concentração de íons Mg2+ tem um grande impacto na eficiência da amplificação por PCR.A concentração excessiva pode reduzir o sexo oposto da amplificação por PCR.Se a concentração for muito baixa, a saída da amplificação do PCR fará até mesmo a falha da amplificação do PCR sem a banda de expansão.
Alteração do volume da reação: O volume usado na amplificação por PCR é 20ul, 30ul e 50ul ou 100uL, o grande volume do aplicativo para amplificação por PCR é definido de acordo com diferentes propósitos de pesquisa científica e testes clínicos.Depois de fazer pequenos volumes como 20ul, é necessário fazer uma condição de cordão ao fazer o tamanho, caso contrário, falhará.
Razões físicas: A transformação é muito importante para a amplificação por PCR.Se a temperatura de degeneração for baixa, o tempo de degeneração for curto, é provável que ocorram falsos negativos;uma temperatura de recozimento muito baixa pode causar amplificação não específica e reduzir a eficiência da amplificação específica.Afeta fortemente a combinação de primers e modelos para reduzir a eficiência da amplificação por PCR.Às vezes é necessário usar termômetros padrão para detectar a variabilidade, recozimento e temperatura estendida na extensão ou fogão solúvel em água, que é uma das razões para a falha do PCR.
Variantes da sequência alvo: Se a sequência alvo ocorrer, uma mutação ou deleção, a combinação do protótipo e do molde for combinada, ou devido à falta de uma sequência alvo, o iniciador e o molde perderão a sequência complementar e sua amplificação por PCR não será bem-sucedida.
● Falso positivo
A banda de amplificação de PCR parece consistente com a banda da sequência alvo e, às vezes, sua banda é mais nítida e alta.
O design do primer não é apropriado: a sequência de amplificação selecionada e a sequência de amplificação sem propósito têm homólogas; portanto, quando a amplificação por PCR, os produtos de PCR amplificados são sequências sem propósito.A sequência alvo é muito curta ou o iniciador é muito curto e é propenso a falsos positivos.Precisa ser redesenhado.
Poluição cruzada da sequência alvo ou produtos de amplificação: Há duas razões para essa poluição: Primeiro, poluição cruzada de todo o genoma ou grandes segmentos, levando a falsos positivos.Esse tipo de falso positivo pode ser resolvido pelos seguintes métodos: Seja cuidadoso e gentil durante a operação para evitar que a sequência de destino seja inalada para a pistola de amostra ou espirre para fora do tubo centrífugo.Com exceção de enzimas e substâncias que não suportam altas temperaturas, todos os reagentes ou equipamentos devem ser desinfetados com alta pressão.Os tubos centrífugos e as amostras devem ser usados ao mesmo tempo.Quando necessário, antes de adicionar amostras, o tubo de reação e o reagente são expostos a raios ultravioleta para destruir o ácido nucléico existente.Segundo, pequenos fragmentos na poluição do ar.Esses pequenos fragmentos são mais curtos que a sequência alvo, mas possuem certa homologia.Pode ser emendado entre si.Após a complementação dos primers, o produto de PCR pode ser expandido, o que ocasionará a produção de falsos positivos.Pode ser usado para reduzir ou eliminar o método de ninho de PCR.
● Aparece banda de amplificação inespecífica
As bandas que apareceram após a amplificação por PCR são inconsistentes com o tamanho esperado, ou grandes ou pequenas, ou ao mesmo tempo, ou ao mesmo tempo, bandas de amplificação específicas e bandas de amplificação não específicas.O surgimento de bandas não específicas é: Primeiro, os primers são complementares incompletos à sequência alvo ou a polimerização do primer para formar um cluster di.A segunda é que a concentração de íons MG2+ é muito alta, a temperatura de recozimento é muito baixa e o número de ciclos de PCR está relacionado.Em segundo lugar, a qualidade e quantidade de enzimas.Freqüentemente, as enzimas de algumas fontes são propensas a bandas não especiais e as enzimas da outra fonte não ocorrem.Às vezes, também ocorre amplificação inespecífica de enzimas.As contramedidas são: atrativos redesenhados, se necessário.Reduza a quantidade de enzima ou substitua a enzima de outra fonte.Reduza a quantidade de primário, aumente a quantidade de modelos adequadamente e reduza o número de ciclos.Aumente adequadamente a temperatura de recozimento ou use o método de dois pontos de temperatura (degeneração de 93°C, recozimento e extensão a cerca de 65°C).
● Aparência de estopa escamosa ou fita adesiva
Às vezes, a amplificação por PCR parece ser aplicada ou com casca ou cinto semelhante a um carpete.Pelo motivo, devido à quantidade excessiva de enzimas ou à má qualidade da enzima, a concentração de dNTP é muito alta, a concentração de Mg2+ é muito alta, a temperatura de recozimento é muito baixa e o número de ciclos é muito grande.As contramedidas são: ①Reduzir a quantidade de enzimas, ou trocar a enzima de outra fonte.②Reduzir a concentração de dNTP ③Reduzir adequadamente a concentração de Mg2+.④Aumente a quantidade de moldes e reduza o número de ciclos.
produtos relacionados
◮ Maior fidelidade: 6 vezes a da enzima Taq comum;
◮ Velocidade de amplificação mais rápida
◮ Mais adaptabilidade do modelo
◮ Maior eficiência de amplificação
◮ A tolerância ambiental é maior: colocado a 37°C por uma semana, mantendo mais de 90% de atividade;
◮ Possui atividade de DNA polimerase 5'→3' e atividade de exonuclease 5'→3', sem atividade de exonuclease 3'→5'.
O sistema de reação exclusivo e a Taq DNA Polimerase de alta eficiência fazem com que a reação de PCR tenha maior eficiência de amplificação, especificidade e sensibilidade.
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
◮ O kit de uma etapa faz transcrição reversa e qPCR duas reações no mesmo tubo, só precisa adicionar RNA modelo, primers de PCR específicos e ddH livre de RNase2O.
◮ O kit pode analisar quantitativamente o RNA viral ou rastrear o RNA de forma rápida e eficiente.
◮ O kit usa um reagente exclusivo de transcrição reversa Foregene e Foregene HotStar Taq DNA Polymerase combinado com um sistema de reação exclusivo para melhorar efetivamente a eficiência da amplificação e a especificidade da reação.
◮ O sistema de reação otimizado faz com que a reação tenha maior sensibilidade de detecção, maior estabilidade térmica e melhor tolerância.
◮ RT-qPCR FácilTM(One Step)-SYBR Green I kit vem com corante de referência interna ROX, que pode ser usado para eliminar sinais de fundo e erros de sinal entre poços, o que é conveniente para os clientes usarem em diferentes modelos de instrumentos quantitativos de PCR.
RT fácilTMII (Pré-mistura principal para síntese de cDNA de primeira cadeia paraPCR em tempo real)
-Capacidade eficiente de remover gDNA, que pode remover gDNA no modelo em 2 minutos.
-Eficiente sistema de transcrição reversa, leva apenas 15 minutos para completar a síntese da primeira fita de cDNA.
-Modelos complexos: modelos com alto conteúdo de GC e estrutura secundária complexa também podem ser revertidos com alta eficiência.
-Sistema de transcrição reversa de alta sensibilidade, os modelos de nível pg também podem obter cDNA de alta qualidade.
-O sistema de transcrição reversa tem alta estabilidade térmica, a temperatura de reação ideal é de 42 ℃ e ainda possui bom desempenho de transcrição reversa a 50 ℃.
Horário da postagem: 18 de março de 2023