Surgimento do SARS-CoV-2 B.1.1.7 Linhagem—
Estados Unidos, 29 de dezembro de 2020–12 de janeiro de 2021
Summer E. Galloway, PhD 1 ;Prabasaj Paul, PhD 1 ;Duncan R. MacCannell, PhD 2 ;Michael A. Johansson, PhD 1 ;
John T. Brooks, MD 1 ;Adam MacNeil, PhD 1 ;Rachel B. Slayton, PhD 1 ;Suxiang Tong, PhD 1 ;Benjamin J. Silk, PhD 1 ;Gregory L. Armstrong, MD 2 ;
Matthew Biggerstaff, ScD 1 ;Vivien G. Dugan, PhD
Em 15 de janeiro de 2021, este relatório foi publicado como um MMWRLiberação antecipada no site do MMWR (https://www.cdc.gov/mmwr).
Em 14 de dezembro de 2020, o Reino Unido informouuma variante preocupante de SARS-CoV-2 (VOC), linhagem B.1.1.7,também referido como VOC 202012/01 ou 20I/501Y.V1.* OEstima-se que a variante B.1.1.7 tenha surgido em setembro2020 e rapidamente se tornou o dominante circulandoVariante do SARS-CoV-2 na Inglaterra (1).B.1.1.7 foidetectado em mais de 30 países, incluindo os Estados Unidos.Comode 13 de janeiro de 2021, aproximadamente 76 casos de B.1.1.7 foramfoi detectado em 12 estados dos EUA.Múltiplas linhas de evidênciaindicam que B.1.1.7 é transmitido com mais eficiência do queoutras variantes do SARS-CoV-2 (1–3).A trajetória modelada deesta variante nos EUA apresenta rápido crescimento no início de 2021,tornando-se a variante predominante em março.AumentouA transmissão do SARS-CoV-2 pode ameaçar os cuidados de saúde sobrecarregadosrecursos, requerem implementação estendida e mais rigorosade estratégias de saúde pública (4), e aumentar a porcentagem deimunidade populacional necessária para o controle da pandemia.Tirandomedidas para reduzir a transmissão agora podem diminuir o potencialimpacto de B.1.1.7 e permitir tempo crítico para aumentar a vacinaçãocobertura de ção.Coletivamente, vigilância genômica aprimoradacombinado com a conformidade contínua com o público eficazmedidas de saúde, incluindo vacinação, distanciamento físico,uso de máscaras, higiene das mãos e isolamento e quarentena,ser essencial para limitar a propagação do SARS-CoV-2, o vírusque causa a doença de coronavírus 2019 (COVID-19).Estratégicotestes de pessoas sem sintomas, mas com maior risco deinfecção, como aqueles expostos a SARS-CoV-2 ou que tenhamcontacto frequente e inevitável com o público, constitui outraoportunidade de limitar a propagação em curso.
Vigilância genômica global e shar rápido de código abertoO processamento de sequências do genoma viral facilitou a análise quase em tempo realdetecção, comparação e rastreamento da evolução do SARS-CoV-2variantes que podem informar os esforços de saúde pública para controlar apandemia.Considerando que algumas mutações no genoma viralsurgem e depois retrocedem, outros podem conferir um avanço seletivoadaptação à variante, incluindo transmissibilidade melhorada, para quetal variante pode dominar rapidamente outras variantes circulantes.
No início da pandemia, variantes do SARS-CoV-2 contendoa mutação D614G na proteína spike (S) que aumentaavidez de ligação ao receptor tornou-se rapidamente dominante em muitosregiões geográficas (5,6).No final do outono de 2020, vários países relataram a detecçãoVariantes do SARS-CoV-2 que se espalham com mais eficiência.Além dissopara a variante B.1.1.7, variantes notáveis incluem o B.1.351linhagem detectada pela primeira vez na África do Sul e a recentemente identificadaB.1.1.28 subclade (renomeado“P.1”) detectado em quatro viajantesdo Brasil durante triagem de rotina no Haneda (Tóquio)aeroporto.§ Essas variantes carregam uma constelação de mutações genéticasções, incluindo no domínio de ligação ao receptor da proteína S,que é essencial para a ligação à angiotensina da célula hospedeiraconvertendo o receptor da enzima-2 (ACE-2) para facilitar o vírusentrada.Evidências sugerem que outras mutações encontradas nessesvariantes podem conferir não apenas maior transmissibilidade, mastambém pode afetar o desempenho de alguns diagnósticos em tempo realTranscrição reversa–reação em cadeia da polimerase (RT-PCR)ensaios¶ e reduzir a suscetibilidade a anticorpos neutralizantes(2,3,5–10).Um relato de caso recente documentou o primeiro caso deReinfecção por SARS-CoV-2 no Brasil com uma variante de SARS-CoV-2que continha a mutação E484K,** que foi demonstradapara reduzir a neutralização por soros convalescentes e monoclonaisanticorpos (9,10).
Este relatório enfoca o surgimento da variante B.1.1.7nos Estados Unidos.A partir de 12 de janeiro de 2021, nem oB.1.351 nem as variantes P.1 foram detectadas noEstados Unidos.Para obter informações sobre o SARS-CoV-2 emergentevariantes de preocupação, o CDC mantém uma página dedicada afornecendo informações sobre variantes emergentes do SARS-CoV-2.††
B.1.1.7 linhagem (20I/501Y.V1)
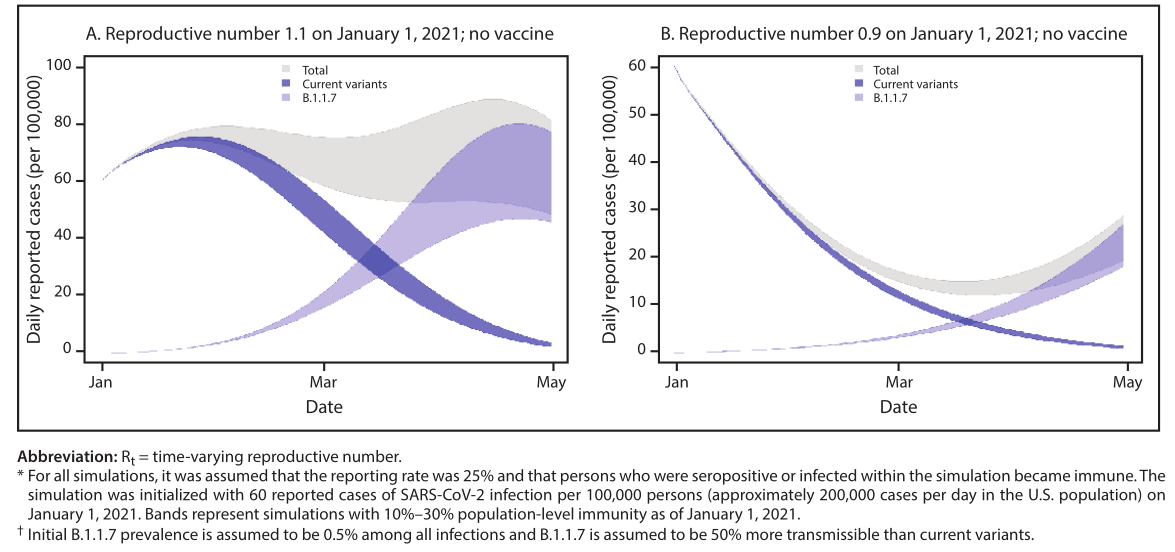
A variante B.1.1.7 carrega uma mutação na proteína S(N501Y) que afeta a conformação de receptores de ligaçãodomínio.Esta variante tem 13 outras mutações definidoras da linhagem B.1.1.7 (Tabela), várias das quais estão na proteína S,incluindo uma exclusão nas posições 69 e 70 (del69–70) queevoluiu espontaneamente em outras variantes do SARS-CoV-2 e éhipótese de aumentar a transmissibilidade (2,7).a deleçãonas posições 69 e 70 causa falha no alvo do gene S (SGTF)em pelo menos um RT-PCR–ensaio de diagnóstico baseado (ou seja, com oEnsaio ThermoFisher Taq Path COVID-19, a variável B.1.1.7formiga e outras variantes com o del69–70 produzem um negativoresultado para o alvo do gene S e um resultado positivo para os outros doismetas);SGTF atuou como procurador no Reino Unidopara identificar casos B.1.1.7 (1).Múltiplas linhas de evidência indicam que B.1.1.7 é maistransmitidos de forma eficiente em comparação com outros SARS-CoV-2variantes que circulam no Reino Unido.regiões do Reino Unido comuma maior proporção de sequências B.1.1.7 teve epidemia mais rápidacrescimento do que em outras áreas, os diagnósticos com SGTF aumentarammais rápido do que os diagnósticos não-SGTF nas mesmas áreas, e ummaior proporção de contatos foram infectados por pacientes índicecom infecções B.1.1.7 do que por pacientes índice infectados comoutras variantes (1,3).A variante B.1.1.7 tem o potencial de aumentar o pan dos EUAtrajetória dêmica nos próximos meses.Para ilustrar esse efeito,um modelo compartimental simples de duas variantes foi desenvolvido.A prevalência atual de B.1.1.7 nos EUA entre todos osvírus é desconhecido, mas acredita-se que seja <0,5% com base nonúmero limitado de casos detectados e dados SGTF (8).Parao modelo, as suposições iniciais incluíam uma prevalência B.1.1.7de 0,5% entre todas as infecções, a imunidade SARS-CoV-2 deinfecção prévia de 10%–30%, um reprodutor variável no temponúmero (R t ) de 1,1 (transmissão atenuada, mas crescente)ou 0,9 (diminuindo a transmissão) para as variantes atuais, e uma incidência relatada de 60 casos por 100.000 pessoas por dia em1º de janeiro de 2021. Essas suposições não representam precisamentequalquer localização única nos EUA, mas sim, indicam uma generalização decondições comuns em todo o país.A mudança em R t ao longotempo decorrente da imunidade adquirida e aumento da prevalênciaência de B.1.1.7, foi modelado, com o B.1.1.7 R t assumidoser uma constante 1,5 vezes o R t das variantes atuais, com base emestimativas iniciais do Reino Unido (1,3).Em seguida, o impacto potencial da vacinação foi modeladoassumindo que 1 milhão de doses de vacina foram administradas pordia começando em 1º de janeiro de 2021, e essa imunidade de 95%foi alcançado 14 dias após o recebimento de 2 doses.Especificamente,imunidade contra a infecção com variantes atuais ou oA variante B.1.1.7 foi assumida, embora a eficácia ea duração da proteção contra a infecção permanece incerta,porque estes não eram o endpoint primário de ensaios clínicospara vacinas iniciais.Neste modelo, a prevalência de B.1.1.7 é inicialmente baixa, mas porqueé mais transmissível do que as variantes atuais, exiberápido crescimento no início de 2021, tornando-se a variável predominanteformiga em março (Figura 1).Se a transmissão de correntevariantes está aumentando (R t inicial = 1,1) ou diminuindo lentamente(R t inicial = 0,9) em janeiro, B.1.1.7 gera uma mudança substancialna trajetória de transmissão e uma nova fase decrescimento.Com a vacinação que protege contra a infecção, otrajetórias epidêmicas iniciais não mudam e B.1.1.7 se espalhaainda ocorre (Figura 2).No entanto, após B.1.1.7 se tornar ovariante dominante, sua transmissão foi substancialmente reduzida.O efeito da vacinação na redução da transmissão no próximoprazo foi maior no cenário em que a transmissão foijá decrescente (R t inicial = 0,9) (Figura 2).Os primeiros esforços quepode limitar a propagação da variante B.1.1.7, como universal emaior conformidade com estratégias de mitigação de saúde pública,permitirá mais tempo para a vacinação em curso para alcançar maiorimunidade em nível populacional.
Discussão
Atualmente, não há diferença conhecida nos resultados clínicosassociado às variantes descritas do SARS-CoV-2;no entanto,uma maior taxa de transmissão levará a mais casos, aumentandoo número total de pessoas que precisam de cuidados clínicos, exacerbarcombatendo o fardo de um sistema de saúde já sobrecarregado,e resultando em mais mortes.Vigilância genômica continuadaidentificar casos B.1.1.7, bem como o surgimento de outrosvariantes de preocupação nos Estados Unidos, é importante para oResposta de saúde pública COVID-19.Considerando que os resultados do SGTFpode ajudar a identificar potenciais B.1.1.7 casos que podem ser confirmadospor sequenciamento, identificando variantes prioritárias que não exibemO SGTF depende exclusivamente da vigilância baseada em sequência.
|
designação variante | Primeira identificação | Mutações características (proteína: mutação) | Nº de casos atuais confirmados por sequência | Nº de países com sequências | ||
| Localização | Data | Estados Unidos | Mundialmente | |||
| B.1.1.7 (20I/501Y.V1) | Reino Unido | Set 2020 | ORF1ab: T1001I, A1708D, I2230T, del3675–3677 SGF S: del69–70 HV, del144 Y, N501Y, A570D, D614G, P681H, T761I, S982A, D1118H ORF8: Q27stop, R52I, Y73C N: D3L, S235F | 76 | 15.369 | 36 |
| B.1.351 (20H/501Y.V2) | África do Sul | outubro de 2020 | ORF1ab: K1655N E: P71L N: T205I S:K417N, E484K, N501Y, D614G, A701V | 0 | 415 | 13
|
| P.1 (20J/501Y.V3 | Brasil e Japão | janeiro de 2021 | ORF1ab: F681L, I760T, S1188L, K1795Q, del3675–3677 SGF, E5662D S: L18F, T20N, P26S, D138Y, R190S, K417T, E484K, N501Y, D614G, H655Y, T1027I ORF3a: C174G ORF8: E92K ORF9: Q77E ORF14: V49L N: P80R | 0 | 35 | 2
|
Abreviações: del = exclusão;E = proteína do envelope;N = proteína do nucleocapsídeo;ORF = quadro de leitura aberto;S = proteína de pico.
A experiência no Reino Unido e os modelos B.1.1.7apresentados neste relatório ilustram o impacto de uma doença mais contagiosavariante pode ter sobre o número de casos em uma população.Oo aumento da transmissibilidade desta variante requer uma abordagem ainda maisimplementação combinada rigorosa de vacinação e mitigaçãomedidas de proteção (por exemplo, distanciamento, mascaramento e higiene das mãos)para controlar a propagação do SARS-CoV-2.Estas medidas serãomais eficazes se forem instituídos mais cedo ou mais tardepara retardar a propagação inicial da variante B.1.1.7.Esforço parapreparar o sistema de saúde para novos surtos de casosgarantido.Maior transmissibilidade também significa que maiordo que a cobertura vacinal prevista deve ser alcançada paraalcançar o mesmo nível de controle de doenças para proteger o públicoem comparação com variantes menos transmissíveis.Em colaboração com instituições acadêmicas, industriais, estaduais, territoriais,parceiros tribais e locais, CDC e outras agências federaisestão coordenando e aprimorando a vigilância genômica eesforços de caracterização de vírus nos Estados Unidos.CDCcoordena os esforços de sequenciamento dos EUA por meio do SARS-CoV-2Sequenciamento para Resposta a Emergências de Saúde Pública,Epidemiologia e Vigilância (SPHERES)§§consórcio,que inclui aproximadamente 170 instituições participantes e promove o compartilhamento aberto de dados para facilitar o uso do SARS-CoV-2dados de sequência.Para rastrear a evolução viral do SARS-CoV-2, o CDC estáimplementando vigilância genômica multifacetada para entenderos processos epidemiológicos, imunológicos e evolutivosque moldam as filogenias virais (filodinâmica);surto de guiainvestigações;e facilitar a detecção e caracterizaçãode possíveis reinfecções, casos de descoberta de vacinas evariantes virais emergentes.Em novembro de 2020, o CDC estabeleceuo Programa Nacional de Vigilância de Cepas de SARS-CoV-2 (NS3)para melhorar a representatividade do SARS-CoV-2 domésticosequências.O programa colabora com 64 instituições públicas dos Estados Unidoslaboratórios de saúde para apoiar um sistema de vigilância genômica;A NS3 também está construindo uma coleção de espécimes SARS-CoV-2nd sequências para apoiar a resposta de saúde pública epesquisa para avaliar o impacto de mutações preocupantes emcontramedidas médicas recomendadas existentes.CDC temtambém contratou vários grandes laboratórios clínicos comerciaistories para sequenciar rapidamente dezenas de milhares de SARS-CoV-2–espécimes positivos a cada mês e financiou sete projetos acadêmicosinstituições para conduzir a vigilância genômica em parceriacom agências de saúde pública, agregando assim substancialmentea disponibilidade de dados de vigilância genômica oportunos de todo oos Estados Unidos.Além dessas iniciativas nacionais,muitas agências de saúde pública estaduais e locais estão sequenciando
FIGURA 1. Trajetórias simuladas de incidência de casos* das variantes atuais do SARS-CoV-2 e da variante B.1.1.7,†supondo que não haja vacinação comunitáriae R t inicial = 1,1 (A) ou R t inicial = 0,9 (B) para variantes atuais—Estados Unidos, janeiro–abril de 2021
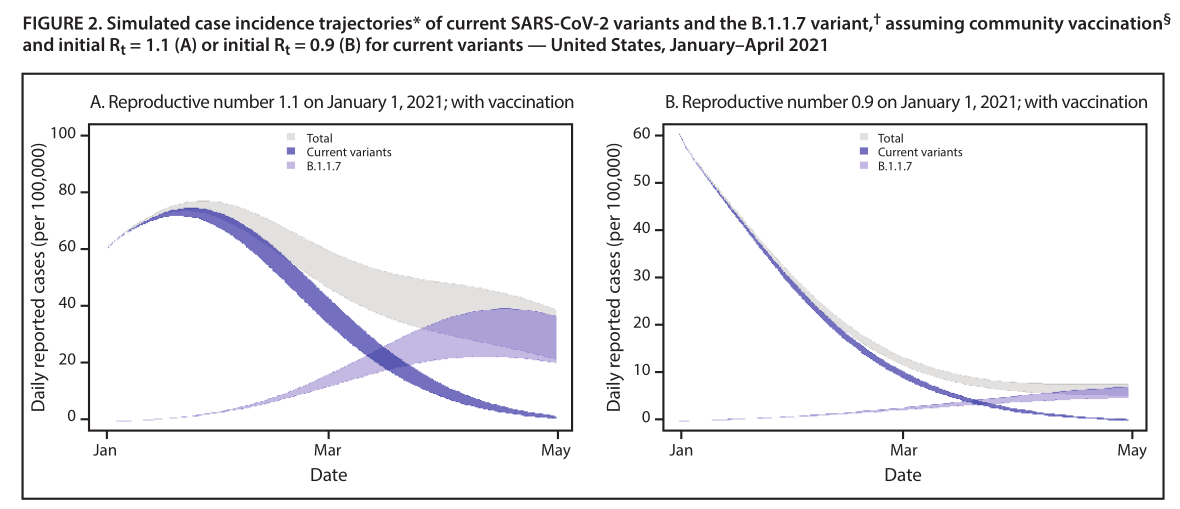


SARS-CoV-2 para entender melhor a epidemiologia local eapoiar a resposta da saúde pública à pandemia.As conclusões deste relatório estão sujeitas a pelo menos três limitestações.Em primeiro lugar, a magnitude do aumento da transmissibilidadenos Estados Unidos em comparação com o observado noReino Unido permanece incerto.Em segundo lugar, a prevalência deB.1.1.7 nos Estados Unidos também é desconhecido neste momento, masdetecção de variantes e estimativa de prevalência melhorarãocom os esforços reforçados de vigilância dos EUA.Finalmente, a mitigação localmedidas de ção também são altamente variáveis, levando a variação emR t .Os resultados específicos apresentados aqui são baseados em simulaçõesções e assumiu nenhuma mudança nas mitigações após 1º de janeiro.O aumento da transmissibilidade da guerra variante B.1.1.7garante a implementação rigorosa de estratégias de saúde pública parareduzir a transmissão e diminuir o impacto potencial de B.1.1.7,ganhando tempo crítico para aumentar a cobertura vacinal.CDCos dados de modelagem mostram que o uso universal e o aumento da conformidadecom medidas de mitigação e vacinação são cruciais parareduzir substancialmente o número de novos casos e mortes nopróximos meses.Além disso, o teste estratégico de pessoas semsintomas de COVID-19, mas que correm maior risco deinfecção com SARS-CoV-2, oferece outra oportunidade paralimitar a propagação em curso.Coletivamente, vigilância genômica aprimoradalance combinado com maior conformidade com a saúde públicaestratégias de mitigação, incluindo vacinação, distanciamento físicoing, uso de máscaras, higiene das mãos e isolamento e quarentena,será essencial para limitar a propagação do SARS-CoV-2 eproteger a saúde pública.
Agradecimentos
Integrantes do Sequenciamento para Emergências em Saúde PúblicaConsórcio de Resposta, Epidemiologia e Vigilância;estadual e locallaboratórios de saúde pública;Associação dos Laboratórios de Saúde Pública;Equipe de Resposta do CDC COVID-19;Divisão de Vírus Respiratórios,Divisão de Doenças Virais, CDC. Formulário do Comitê de Editores de Revistas Médicas para divulgação de potencialconflitos de interesse.Não foram divulgados potenciais conflitos de interesse.
Referências
1. Saúde Pública da Inglaterra.Investigação da nova variante do SARS-CoV-2: variante preocupante 202012/01, resumo técnico 3. Londres, Reino Unido: Public Health England;2020. https://assets.publishing.service.gov.uk/government/uploads/system/uploads/attachment_data/file/950823/Variant_of_Concern_VOC_202012_01_Technical_Briefing_3_-_England.pdf
2. Kemp SA, Harvey WT, Datir RP, et al.Surgimento e transmissão recorrentes de uma deleção de pico SARS-CoV-2 ΔH69/V70.bioRxiv[Pré-impressão publicada online em 14 de janeiro de 2021].https://www.biorxiv.org/content/10.1101/2020.12.14.422555v4
3. Volz E, Mishra S, Chand M, et al.Transmissão da linhagem B.1.1.7 do SARS-CoV-2 na Inglaterra: insights da associação de dados epidemiológicos e genéticos.medRxiv [Pré-impressão publicada online em 4 de janeiro de 2021].https://www.medrxiv.org/content/10.1101/2020.12.30.20249034v2
4. Honein MA, Christie A, Rose DA, e outros;Equipe de resposta do CDC COVID-19.Resumo das orientações para estratégias de saúde pública para lidar com altos níveis de transmissão comunitária de SARS-CoV-2 e mortes relacionadas, dezembro de 2020. MMWR Morb Mortal Wkly Rep 2020;69:1860–7.PMID:33301434 https://doi.org/10.15585/mmwr.mm6949e2
5. Volz E, Hill V, McCrone JT, e outros;Consórcio COG-Reino Unido.Avaliando os efeitos da mutação de pico SARS-CoV-2 D614G na transmissibilidade e patogenicidade.Célula 2021;184:64–75.e11.PMID:33275900 https://doi.org/10.1016/j.cell.2020.11.020
6. Korber B, Fischer WM, Gnanakaran S, e outros;Sheffield COVID-19 Genomics Group.Alterações de rastreamento no pico de SARS-CoV-2: evidências de que o D614G aumenta a infectividade do vírus COVID-19.Célula
2020;182:812–27.PMID:32697968 https://doi.org/10.1016/j.cell.2020.06.043
7. McCarthy KR, Rennick LJ, Namnulli S, et al.Deleções naturais no escapamento de anticorpo de condução de glicoproteína de pico SARS-CoV-2.bioRxiv [Pré-impressão publicada online em 19 de novembro de 2020].https://www.biorxiv.org/content/
10.1101/2020.11.19.389916v18.Washington NL, White S, Schiabor KM, Cirulli ET, Bolze A, Lu JT.S padrões de abandono de genes em testes SARS-CoV-2 sugerem a disseminação da mutação H69del/V70del nos EUA.medRxiv [Pré-impressão publicada online em 30 de dezembro de 2020].https://www.medrxiv.org/content/10.1101/2020.12.24.20248814v1
9. Weisblum Y, Schmidt F, Zhang F, et al.Fuja dos anticorpos neutralizantes por variantes da proteína spike SARS-CoV-2.eLife 2020;9:e61312.PMID:33112236 https://doi.org/10.7554/eLife.61312
10. Greaney AJ, Loes AN, Crawford KHD, et al.Mapeamento abrangente de mutações no domínio de ligação ao receptor SARS-CoV-2 que afetam o reconhecimento por anticorpos séricos humanos policlonais.bioRxiv [Pré-impressão publicada online em 4 de janeiro de 2021].https://www.biorxiv.org/content/10.1101/2020.12.31.425021v1
Horário de postagem: 11 de fevereiro de 2021








