1. Conhecimento básico (se quiser ver a parte experimental, por favor, transfira diretamente para a segunda parte)
Como uma reação derivada da PCR convencional, a PCR em tempo real monitora principalmente a alteração da quantidade de produto de amplificação em cada ciclo da reação de amplificação da PCR em tempo real através da alteração do sinal de fluorescência e analisa quantitativamente o modelo inicial através da relação entre o valor ct e a curva padrão.
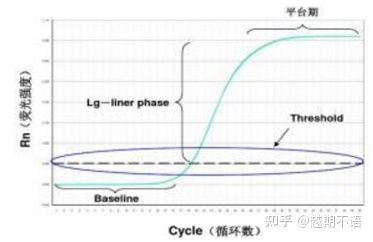
Os dados específicos de RT-PCR sãolinha de base, limiar de fluorescênciaevalor Ct.
| linha de base: | O valor de fluorescência do 3º-15º ciclo é a linha de base (linha de base), que é causada pelo erro ocasional da medição. |
| Limiar (limiar): | Refere-se ao limite de detecção de fluorescência definido em uma posição apropriada na região de crescimento exponencial da curva de amplificação, geralmente 10 vezes o desvio padrão da linha de base. |
| valor CT: | É o número de ciclos de PCR quando o valor de fluorescência em cada tubo de reação atinge o limite. O valor Ct é inversamente proporcional à quantidade de molde inicial. |
Métodos de rotulagem comuns para RT-PCR:
| método | vantagem | deficiência | âmbito de aplicação |
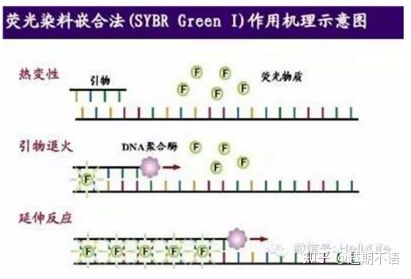
| SYBR VerdeⅠ | Ampla aplicabilidade, sensível, barato e conveniente | Os requisitos de primer são altos, propensos a bandas não específicas | É adequado para análise quantitativa de vários genes-alvo, pesquisa sobre expressão gênica e pesquisa em animais e plantas transgênicos recombinantes. |
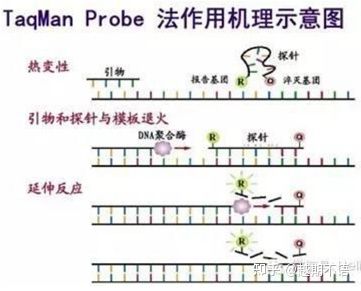
| TaqManName | Boa especificidade e alta repetibilidade | O preço é alto e adequado apenas para objetivos específicos. | Detecção de patógenos, pesquisa de genes de resistência a medicamentos, avaliação da eficácia de medicamentos, diagnóstico de doenças genéticas. |
| farol molecular | Alta especificidade, fluorescência, fundo baixo | O preço é alto, é adequado apenas para uma finalidade específica, o design é difícil e o preço é alto. | Análise genética específica, análise SNP |
2. Etapas experimentais
2.1 Sobre o agrupamento experimental- deve haver múltiplos poços no grupo, e deve haver repetições biológicas.
| ① | controle em branco | Usado para detectar o status de crescimento celular em experimentos |
| ② | SiRNA de controle negativo (sequência de siRNA não específica) | Demonstrar a especificidade da ação do RNAi.O siRNA pode induzir uma resposta de estresse não específica a uma concentração de 200 nM. |
| ③ | Controle de Reagente de Transfecção | Excluir a toxicidade do reagente de transfecção para as células ou o efeito na expressão do gene alvo |
| ④ | siRNA contra o gene alvo | Derrubar a expressão do gene alvo |
| ⑤ (opcional) | siRNA positivo | Usado para solucionar problemas do sistema experimental e problemas operacionais |
| ⑥ (opcional) | siRNA de controle fluorescente | A eficiência da transfecção celular pode ser observada com um microscópio |
2.2 Princípios de design de primer
| Tamanho do fragmento amplificado | De preferência em 100-150bp |
| Comprimento do primer | 18-25bp |
| conteúdo do GC | 30%-70%, preferencialmente 45%-55% |
| valor tm | 58-60 ℃ |
| Seqüência | Evite T/C contínuo;A/G contínua |
| 3 sequência final | Evite os ricos em GC ou AT;a base terminal é preferencialmente G ou C;é melhor evitar T |
| Complementaridade | Evite sequências complementares de mais de 3 bases dentro do primer ou entre dois primers |
| Especificidade | Use a pesquisa de explosão para confirmar a especificidade do primer |
①SiRNA é específico da espécie, e as sequências de diferentes espécies serão diferentes.
②SiRNA é embalado em pó liofilizado, que pode ser armazenado de forma estável por 2-4 semanas em temperatura ambiente.
2.3 Ferramentas ou reagentes que precisam ser preparados com antecedência
| Primer (referência interna) | Incluindo para frente e para trás dois |
| Primers (gene alvo) | Incluindo para frente e para trás dois |
| Alvo Si RNA (3 tiras) | Geralmente, a empresa sintetiza 3 tiras e depois escolhe uma das três por RT-PCR |
| Kit de Transfecção | Lipo2000 etc |
| Kit de extração rápida de RNA | Para extração de RNA após transfecção |
| Kit de Transcrição Reversa Rápida | para síntese de cDNA |
| Kit de amplificação de PCR | 2×Super SYBR Verde qPCR Master Mix |
2.4 Em relação às questões que precisam ser observadas nas etapas experimentais específicas:
①processo de transfecção de siRNA
1. Para plaqueamento, você pode escolher placa de 24 poços, placa de 12 poços ou placa de 6 poços (a concentração média de RNA proposta em cada poço de uma placa de 24 poços é de cerca de 100-300 ng/uL), e a densidade ideal de transfecção de células é de até 60%-80% ou mais
2. As etapas de transfecção e requisitos específicos estão estritamente de acordo com as instruções.
3. Após a transfecção, as amostras podem ser coletadas dentro de 24-72 horas para detecção de mRNA (RT-PCR) ou detecção de proteína dentro de 48-96 horas (WB)
② Processo de extração de RNA
1. Evitar a contaminação por enzimas exógenas.Inclui principalmente o uso estrito de máscaras e luvas;usando pontas de pipeta esterilizadas e tubos EP;a água utilizada no experimento deve ser livre de RNase.
2. Recomenda-se fazer o dobro do sugerido no kit de extração rápida, o que realmente melhorará a pureza e o rendimento.
3. O líquido residual não deve tocar na coluna de RNA.
③ Quantificação de RNA
Depois que o RNA é extraído, ele pode ser quantificado diretamente com Nanodrop, e a leitura mínima pode ser tão baixa quanto 10 ng/ul.
④Processo de transcrição reversa
1. Devido à alta sensibilidade do RT-qPCR, pelo menos 3 poços paralelos devem ser feitos para cada amostra para evitar que o Ct subseqüente seja muito diferente ou o SD seja muito grande para análise estatística.
2. Não congele e descongele a mistura principal repetidamente.
3. Cada tubo/orifício deve ser substituído por uma nova ponta!Não use continuamente a mesma ponta de pipeta para adicionar amostras!
4. O filme anexado à placa de 96 poços após a adição da amostra precisa ser alisado com uma placa.É melhor centrifugar antes de colocar na máquina, para que o líquido na parede do tubo possa escorrer e remover as bolhas de ar.
⑤Análise de curva comum
| Sem período de crescimento logarítmico | Possivelmente alta concentração de modelo |
| Sem valor CT | Etapas incorretas para detecção de sinais fluorescentes; degradação de primers ou sondas – sua integridade pode ser detectada por eletroforese PAGE; quantidade insuficiente de gabarito; degradação de templates – evitando a introdução de impurezas e repetidos congelamentos e descongelamentos no preparo de amostras; |
| Ct>38 | Baixa eficiência de amplificação;O produto de PCR é muito longo;vários componentes da reação são degradados |
| Curva de amplificação linear | As sondas podem ser parcialmente degradadas por ciclos repetidos de congelamento e descongelamento ou exposição prolongada à luz |
| A diferença em furos duplicados é particularmente grande | A solução de reação não está completamente fundida ou a solução de reação não está misturada;o banho térmico do instrumento de PCR está contaminado por substâncias fluorescentes |
2.5 Sobre análise de dados
A análise de dados de qPCR pode ser dividida em quantificação relativa e quantificação absoluta.Por exemplo, células do grupo de tratamento comparadas com células do grupo de controle,
Quantas vezes o mRNA do gene X muda, isso é uma quantificação relativa;em um certo número de células, o mRNA do gene X
Quantas cópias existem, isso é quantificação absoluta.Normalmente o que mais usamos no laboratório é o método quantitativo relativo.Geralmente,o método 2-ΔΔcté mais usado em experimentos, então apenas este método será apresentado em detalhes aqui.
Método 2-ΔΔct: O resultado obtido é a diferença na expressão do gene alvo no grupo experimental em relação ao gene alvo no grupo controle.É necessário que as eficiências de amplificação do gene alvo e do gene de referência interno sejam próximas de 100% e o desvio relativo não exceda 5%.
O método de cálculo é o seguinte:
Grupo de controle Δct = valor ct do gene alvo no grupo de controle - valor ct do gene de referência interno no grupo de controle
Δct grupo experimental = valor ct do gene alvo no grupo experimental – valor ct do gene de referência interna no grupo experimental
ΔΔct=Δct grupo experimental-Δct grupo de controle
Por fim, calcule o múltiplo da diferença no nível da expressão:
Change Fold=2-ΔΔct (correspondente à função excel é POWER)
Produtos relacionados:
Horário de postagem: 20 de maio de 2023