Material de partida: ARN
A PCR quantitativa de transcrição reversa (RT-qPCR) é um método experimental usado em experimentos de PCR usando RNA como material de partida.Neste método, o RNA total ou RNA mensageiro (mRNA) é primeiro transcrito em DNA complementar (cDNA) pela transcriptase reversa.Posteriormente, uma reação de qPCR foi realizada usando o cDNA como modelo.RT-qPCR tem sido usado em uma variedade de aplicações de biologia molecular, incluindo análise de expressão gênica, validação de interferência de RNA, validação de microarray, detecção de patógenos, testes genéticos e pesquisa de doenças.
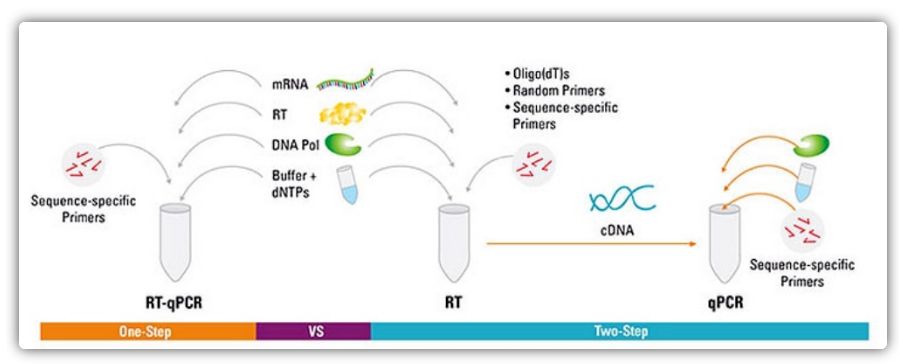
Métodos de uma e duas etapas para RT-qPCR
RT-qPCR pode ser realizado por um método de uma ou duas etapas.A RT-qPCR de uma etapa combina a transcrição reversa e a amplificação por PCR, permitindo que a transcriptase reversa e a DNA polimerase concluam a reação no mesmo tubo sob as mesmas condições de tampão.A RT-qPCR de uma etapa requer apenas o uso de primers específicos de sequência.Na RT-qPCR de duas etapas, a transcrição reversa e a amplificação por PCR são realizadas em dois tubos, usando diferentes tampões otimizados, condições de reação e estratégias de design de primer.
| Vantagem | Desvantagem | |
| Um passo | Este método tem menos erro experimental, pois ambas as reações são feitas em um tubo
Menos etapas de pipetagem reduzem o risco de contaminação
Adequado para amplificação/triagem de alto rendimento, rápido e reprodutível | Reações em duas etapas não podem ser otimizadas separadamente
Como as condições de reação são comprometidas pela combinação da reação em duas etapas, a sensibilidade não é tão boa quanto a do método de duas etapas
O número de alvos detectados por uma única amostra é pequeno |
| Dois passos | Capacidade de criar bibliotecas de cDNA estáveis que podem ser armazenadas por longos períodos de tempo e usadas em múltiplas reações
Os genes-alvo e os genes de referência podem ser amplificados da mesma biblioteca de cDNA sem a necessidade de várias bibliotecas de cDNA
Tampões de reação e condições de reação que permitem a otimização de execuções de reação única
Seleção flexível de condições de disparo | O uso de vários tubos e mais etapas de pipetagem aumenta o risco de contaminação do DNA, e demorado.
Requer mais otimização do que o método de uma etapa |
Produtos relacionados:
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
RT-qPCR Easyᵀᴹ (One Step)-Taqman
RT Easyᵀᴹ I Master Premix para síntese de CDNA de primeira cadeia
Kit de PCR em tempo real Easyᵀᴹ-SYBR Green I
PCR em tempo real Easyᵀᴹ-Taqman
Seleção de RNA total e mRNA
Ao projetar um experimento de RT-qPCR, é importante decidir se deve usar RNA total ou mRNA purificado como modelo para a transcrição reversa.Embora o mRNA possa fornecer uma sensibilidade ligeiramente maior, o RNA total ainda é usado com frequência.A razão para isso é que o RNA total tem uma vantagem mais importante como material de partida do que o mRNA.Primeiro, o processo requer menos etapas de purificação, o que garante melhor recuperação quantitativa do modelo e melhor normalização dos resultados para números de células iniciais.Em segundo lugar, evita a etapa de enriquecimento de mRNA, que pode evitar a possibilidade de resultados distorcidos devido a diferentes recuperações de diferentes mRNAs.No geral, uma vez que na maioria das aplicações a quantificação relativa do gene alvo é mais importante do que a sensibilidade absoluta da detecção, o RNA total é mais adequado na maioria dos casos.
Iniciador de transcrição reversa
No método de duas etapas, três métodos diferentes podem ser usados para iniciar a reação do cDNA: iniciadores oligo(dT), iniciadores aleatórios ou iniciadores específicos de sequência.Normalmente, primers oligo(dT) e primers aleatórios são usados em combinação.Esses primers se ligam à fita molde do mRNA e fornecem à transcriptase reversa um ponto de partida para a síntese.
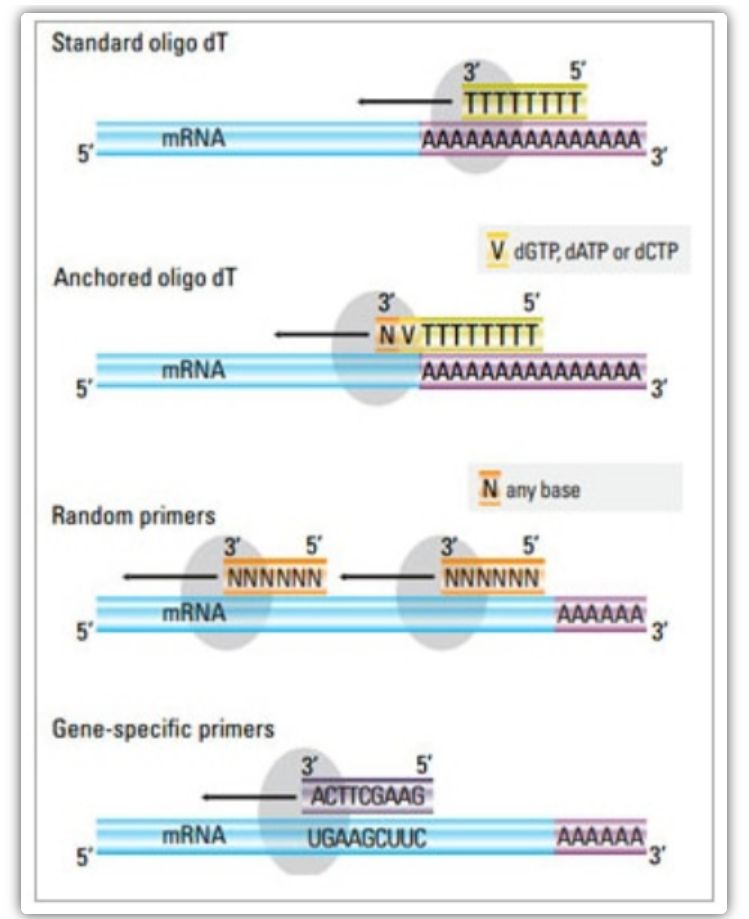
| Seleção de primer | Estrutura e função | Vantagem | Desvantagem |
| Primário oligo(dT) (ou iniciador oligo(dT) ancorado) | Recozimento estendido para resíduos de timina na cauda poli(A) do mRNA;O primer oligo(dT) âncora contém um G, C ou A na extremidade 3' (sítio âncora) | Síntese de cDNA de comprimento total a partir de mRNA de cauda poli(A)
Aplicável quando menos material de partida está disponível
O local de ancoragem garante que o primer oligo(dT) se ligue à cauda poli(A) 5' do mRNA | Adequado apenas para amplificar genes com caudas poli(A)
Obtenha o cDNA truncado do local de priming*2 em poli(A)
Polarizado para se ligar à extremidade 3'*
*Essa possibilidade é minimizada se forem usados primers oligo(dT) ancorados |
| iniciador aleatório
| 6 a 9 bases de comprimento, que podem emparelhar em vários locais durante a transcrição do RNA | Recozimento para todos os RNAs (tRNA, rRNA e mRNA)
Adequado para transcritos com estrutura secundária significativa ou quando menos material de partida está disponível
Alto Rendimento de cDNA | O cDNA é transcrito reversa de todo o RNA, o que geralmente não é desejado e pode diluir o sinal do mRNA alvo
obter cDNA truncado |
| primers específicos de sequência | Iniciadores personalizados direcionados a sequências específicas de mRNA | biblioteca de cDNA específica
Melhorar a sensibilidade
Usando iniciadores de qPCR reverso | Apenas limitado à síntese de um único gene alvo |
Transcriptase reversa
A transcriptase reversa é uma enzima que usa o RNA para sintetizar o DNA.Algumas transcriptases reversas têm atividade de RNase e podem degradar fitas de RNA em fitas híbridas de RNA-DNA após a transcrição.Se não tiver atividade enzimática RNase, RNaseH pode ser adicionado para maior eficiência de qPCR.As enzimas comumente usadas incluem a transcriptase reversa do vírus da leucemia murina de Moloney e a transcriptase reversa do vírus do mieloblastoma aviário.Para RT-qPCR, é ideal escolher uma transcriptase reversa com maior termoestabilidade, para que a síntese de cDNA possa ser realizada em temperaturas mais altas, garantindo a transcrição bem-sucedida de RNAs com maior estrutura secundária, mantendo sua atividade plena ao longo da reação, resultando em maiores rendimentos de cDNA.
Produtos relacionados:
Transcriptase Reversa Foreasy M-MLV
Atividade de RNase H da transcriptase reversa
RNaseH é capaz de degradar fitas de RNA de duplexes RNA-DNA, permitindo a síntese eficiente de DNA de fita dupla.No entanto, ao usar mRNA longo como modelo, o RNA pode ser degradado prematuramente, resultando em cDNA truncado.Portanto, muitas vezes é benéfico minimizar a atividade de RNaseH durante a clonagem de cDNA se a síntese de transcritos longos for desejada.Em contraste, as transcriptases reversas com atividade de RNase H são frequentemente benéficas para aplicações qPCR porque aumentam a fusão de duplexes RNA-DNA durante o primeiro ciclo de PCR.
Projeto de primer
Os primers de PCR usados para a etapa qPCR em RT-qPCR devem idealmente ser projetados para abranger uma junção exon-exon, onde um primer de amplificação poderia potencialmente abranger um limite real de exon-intron.Como as sequências de DNA genômico contendo íntrons não são amplificadas, esse design reduz o risco de falsos positivos amplificados por DNA genômico contaminante.
Se os primers não puderem ser projetados para separar éxons ou limites de éxon-éxon, pode ser necessário tratar amostras de RNA com DNase I ou dsDNase livre de RNase para remover a contaminação do DNA genômico.
Controle de RT-qPCR
Um controle negativo de transcrição reversa (controle -RT) deve ser incluído em todos os experimentos de RT-qPCR para detectar contaminação de DNA (como DNA genômico ou produtos de PCR de reações anteriores).Este controle contém todos os componentes da reação, exceto a transcriptase reversa.Uma vez que a transcrição reversa não ocorre com este controle, se a amplificação por PCR for observada, a contaminação do DNA é mais provável.
Horário de postagem: 02 de agosto de 2022








