1. Detectar a absorbância da solução de RNA
A absorbância a 280, 320, 230 e 260 nm representa os valores de ácido nucleico, fundo (turbidez da solução), concentração de sal e matéria orgânica, como proteína, respectivamente.Geralmente, olhe apenas para OD260/OD280 (Ratio, R).Quando 1,8~2,0, pensamos que a contaminação de proteína ou outra matéria orgânica no RNA pode ser tolerada, mas deve-se notar que quando Tris é usado como tampão para detectar a absorbância, o valor R pode ser maior que 2 (geralmente deve ser <2,2).Quando R <1,8, a poluição de proteína ou outra matéria orgânica na solução é mais óbvia, e o destino do RNA pode ser determinado de acordo com as necessidades.Quando R>2,2, significa que o RNA foi hidrolisado em um único ácido nucleico.
2.Padrão eletroforético de RNA
Geralmente, o gel desnaturante é usado para eletroforese de RNA, mas se for apenas para detectar a qualidade do RNA, o gel desnaturante não é necessário e o gel de agarose comum pode ser usado.O objetivo da eletroforese é detectar a integridade das bandas 28S e 18S e sua proporção, ou a integridade do esfregaço de mRNA.Geralmente, se as bandas 28S e 18S são brilhantes, claras e nítidas (referindo-se às bordas das bandas claras) e o brilho de 28S é mais que o dobro da banda 18S, consideramos a qualidade do RNA boa.
Os dois métodos acima são comumente usados, mas nenhum desses dois métodos pode nos dizer claramente se há RNase residual na solução de RNA.Se houver uma quantidade muito pequena de RNase na solução, é difícil para nós detectá-la com o método acima, mas a maioria das reações enzimáticas subsequentes são realizadas acima de 37 graus e por um longo tempo.Dessa forma, se houver uma quantidade muito pequena de RNase na solução de RNA, haverá um ambiente e tempo muito adequados para desempenhar seu papel nos experimentos subsequentes e, claro, o experimento estará frio neste momento.Abaixo apresentamos um método que pode confirmar se há RNase residual na solução de RNA.
3. Teste de preservação de calor
De acordo com a concentração da amostra, retire dois RNA de 1000 ng da solução de RNA e adicione-o a um tubo de centrífuga de 0,5 ml e complemente-o com tampão Tris pH 7,0 até um volume total de 10 ul e, em seguida, sele a tampa do tubo.Colocar um deles em banho-maria de temperatura constante a 70°C e mantê-lo aquecido por 1 h.A outra parte foi armazenada em geladeira a -20°C por 1 hora.Quando o tempo acabar, remova as duas amostras para eletroforese.Após a conclusão da eletroforese, compare as bandas eletroforéticas dos dois.Se as bandas dos dois forem consistentes ou não tiverem diferença significativa (claro, suas bandas também atendem às condições do método 2), isso significa que não há contaminação residual de RNase na solução de RNA e a qualidade do RNA é muito boa.Pelo contrário, se a amostra incubada a 70°C apresentar degradação óbvia, isso indica que há contaminação de RNase na solução de RNA.
2 Métodos e técnicas experimentais para extração de RNA
Os problemas que frequentemente encontramos ao extrair o RNA são: (1) o rendimento do RNA é baixo;(2) o RNA tem poluição salina grave;(3) o RNA tem poluição grave de solvente orgânico;(4) degradação da amostra e outros problemas
1. Reagentes de extração de RNA total comumente usados
O método do isotiocianato de guanidina e o método Trizol são os métodos mais comumente usados para a extração de RNA total de tecidos e células animais.É especialmente adequado para pequenas amostras e tecidos que são particularmente difíceis de extrair, como a extração de RNA total de pele de coelho e tecido conjuntivo animal;além disso, o Trizol, como reagente de lise de uso geral, também pode ser usado para a extração de tecidos vegetais, bactérias, fungos e outros tecidos.Para tecidos vegetais contendo polissacarídeos e polifenóis, como camélia oleífera, folhas de chá, colza, etc., o método CTAB também pode ser usado para extrair o RNA total.
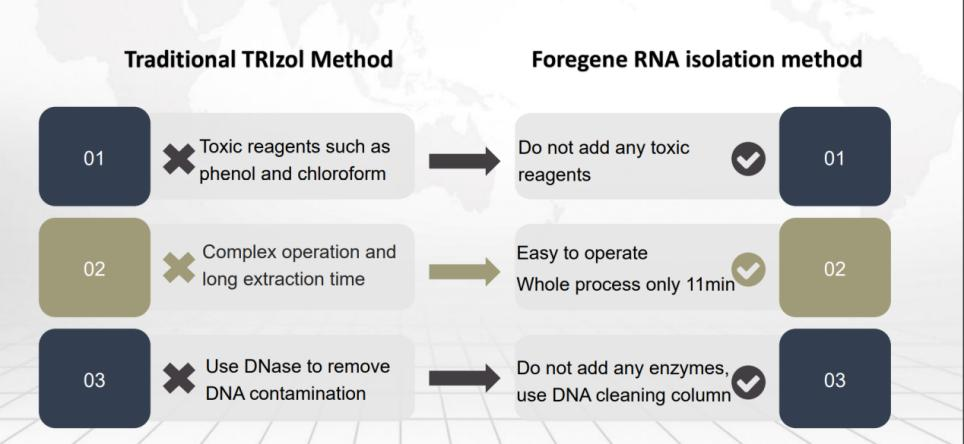
Como um método convencional, o método de coluna dupla também é muito popular devido à sua operação de temperatura normal, sem necessidade de adicionar RNase e segurança - sem clorofórmio, fenóis e outros reagentes orgânicos para extração.(produtos recomendados )

2. Extração de RNA total de tecidos animais
(1) Tente escolher tecido fresco, se não for fresco (de preferência dentro de três meses - 80 ℃ geladeira ou congelado em nitrogênio líquido. Ao cortar tecido, não corte diretamente à temperatura ambiente, certifique-se de colocá-lo na caixa de gelo, tente evitar congelamento e descongelamento repetidos.
(2) Use tesouras e pinças limpas para cortar um pequeno pedaço de tecido, tente cortar a parte central do tecido ao cortar a amostra ou primeiro corte o pedaço grande de tecido do meio e depois corte a amostra na posição de incisão fresca.O tecido removido deve ser totalmente fragmentado, coloque o tecido fragmentado em um tubo EP sem RNase, adicione o lisado, o tecido fragmentado deve ser totalmente exposto ao lisado e prepare-se para a homogeneização.
(3) Para tecidos normais, selecione tecidos do tamanho de feijão mungo (30-60 mg) para homogeneização.Se os tecidos contiverem uma grande quantidade de proteína, gordura ou tecidos fibrosos densos, como o fígado, aumente ou diminua adequadamente a quantidade de tecidos cortados (opcional) Escolha 10 a 20 mg).
(4) Se músculo de peixe, carne de camarão, água-viva e outros tecidos com alto teor de água forem extraídos, o volume da amostra deve ser aumentado adequadamente (recomendado 100-200 mg).
(5) Se as condições permitirem, o tecido animal pode ser extraído diretamente após ser homogeneizado com um homogeneizador de tecido de alta passagem, se não houver tal equipamento.
(6) O RNA obtido após a extração final deve ser colocado na caixa de gelo imediatamente para reduzir a degradação do RNA.
3. Extração de RNA de células animais
(1) Células em suspensão: centrifugar diretamente e descartar o meio, lavar com PBS estéril por 1-2 vezes, suspender com uma quantidade apropriada de PBS e adicionar lisado para lise.Não adicione o lisado diretamente às células precipitadas após descartar completamente o líquido.Isso fará com que o pacote de histonas liberado após as células lisadas na camada externa adira ao exterior das células precipitadas, limitando assim o contato das células dentro do sedimento com o lisado., resultando em lise celular incompleta e produção de RNA reduzida.
(2) Células que são semi-aderentes ou não fortemente aderentes: Após descartar o meio, lave com PBS por 1-2 vezes, então absorva diretamente uma quantidade apropriada de PBS e sopre a placa de cultura com uma pipeta ou pistola para remover as células e transfira-as para células livres de RNA.Adicione o lisado ao tubo EP da enzima para extração.
(3) Células aderentes: precisam ser digeridas com tripsina primeiro, depois coletadas em tubos EP livres de RNase, centrifugadas para remover o sobrenadante, lavadas 1-2 vezes com PBS para remover o excesso de tripsina e ressuspensas com uma quantidade apropriada de PBS Em seguida, prossiga para a etapa de extração.
4. Extração de RNA vegetal
Os tecidos vegetais são ricos em compostos fenólicos, ou ricos em polissacarídeos, ou contêm alguns metabólitos secundários não identificados, ou possuem alta atividade de RNase.Essas substâncias são fortemente combinadas com o RNA após a lise celular para formar complexos insolúveis ou precipitados coloidais, que são difíceis de remover.Portanto, quando extraímos tecido vegetal, precisamos escolher um kit para plantas.O lisado no kit pode efetivamente resolver os problemas de fácil oxidação de polifenóis e separação de compostos polissacarídeos e ácidos nucléicos.
(Para extração de RNA de planta de polissacarídeo polifenol, produtos recomendados:
(1) A casca, polpa, sementes, folhas, etc. da planta devem ser totalmente moídas em pilão.Durante o processo de moagem, o nitrogênio líquido deve ser reabastecido a tempo para evitar o derretimento da amostra.A amostra moída deve ser rapidamente adicionada ao lisado e agitada para evitar a degradação do RNA.
(2) Para amostras ricas em fibras, como arroz e folhas de trigo, a quantidade de extração deve ser reduzida adequadamente, caso contrário, a moagem e a lise do tecido não serão completas, resultando em um baixo rendimento de RNA extraído.
(3) Para tecidos vegetais com alto teor de água, como frutas de romã, frutas de melancia, frutas de pêssego, etc., o tamanho da amostra deve ser aumentado adequadamente (100-200 mg é opcional).
(4) Tecidos vegetais, como folhas de plantas, rizomas, frutas duras e outros materiais são geralmente recomendados para usar nitrogênio líquido para argamassar completamente os ingredientes em um almofariz e, em seguida, prosseguir para a etapa de extração.Os homogeneizadores de tecido convencionais podem não ser eficazes na homogeneização de tecidos vegetais e geralmente não são recomendados.
5. Precauções para extração de RNA
(1) As amostras de tecido devem ser tão frescas quanto possível para evitar congelamentos e descongelamentos repetidos.
(2) O tecido deve ser totalmente triturado durante a extração, e a quantidade de tecido não deve ser muito pequena, muito menos muito.
(3) Deve ser dado tempo de incubação suficiente após a adição do lisado para lisar totalmente a amostra.
(4) Ao usar o método Trizol para extração, o princípio de absorção do sobrenadante após a estratificação é "prefira inalar menos do que inalar mais", e não deve extrair até a camada intermediária, caso contrário, causará séria contaminação do DNA genômico.
(5) Ao lavar, o líquido de lavagem deve se infiltrar totalmente ao redor da parede do tubo para garantir uma lavagem completa.
(6) Para o método de extração em coluna, além de separar a coluna após a lavagem, a coluna de adsorção também deve ser colocada em uma bancada ultralimpa e soprada por 5 a 10 minutos para evaporar totalmente o solvente orgânico até a secura.
(7) Na última eluição do método da coluna, após adicionar água DEPC, ela deve ser incubada por 3-5 minutos, ou a água DEPC deve ser aquecida a 60°C com antecedência para aumentar o rendimento da eluição.No método tradicional de clivagem de Trizol e precipitação de isopropanol, o RNA final é dissolvido em água DEPC, então um tempo apropriado deve ser dado para a dissolução, e o fundo do tubo de centrífuga deve ser soprado continuamente com uma ponta de pipeta.
3 TTrês causas e soluções para baixa concentração de RNA/má qualidade
1. O rendimento é muito baixo
A amostra extraída é muito baixa, a quantidade total é insuficiente ou a amostra extraída é demais e a lise não está completa;o tecido ou células de qualidade apropriada devem ser usados para extração, o pré-tratamento da amostra deve ser bem feito e a lise deve ser suficiente.
2. Resíduos do genoma
Ao extrair pelo método Trizol, quando o sobrenadante é sugado para a camada intermediária após a estratificação, será causada uma séria contaminação do genoma;cuidado extra deve ser tomado ao colocar em camadas para evitar a sucção na camada do meio.Se o método de coluna for usado para extração, um kit contendo DNase I pode ser selecionado para extração.O ácido nucleico adsorvido na membrana é digerido diretamente com DNase I, o que pode reduzir bastante os resíduos de DNA.
3. Degradação de RNA
Pode ser a degradação da própria amostra extraída ou a degradação causada durante o processo de extração;na medida do possível, amostras frescas devem ser usadas para extração de RNA, e as amostras coletadas devem ser armazenadas em nitrogênio líquido ou -80°C na geladeira a tempo, e o congelamento e descongelamento repetidos devem ser evitados.Pontas livres de RNase/DNase, tubos de centrífuga e outros materiais devem ser usados no processo de extração de RNA.O processo de extração deve ser o mais rápido possível.O RNA extraído deve ser colocado em uma caixa de gelo e armazenado a -80 no tempo.Se o RNA extraído precisar ser detectado por eletroforese em gel, a eletroforese deve ser realizada imediatamente após a extração e o tampão de eletroforese deve ser substituído por um recém-preparado.
4. Sal e resíduos de solventes orgânicos
Os reagentes de extração contêm sais de fenol e guanidina e a solução de lavagem contém etanol.Durante o processo de extração, o lisado não foi completamente absorvido e descartado, e a solução de lavagem não foi totalmente seca.Os sais residuais e os solventes orgânicos são prejudiciais à transcrição reversa e à PCR subsequentes.Diferentes graus de inibição, de modo que o lisado tecidual deve ser totalmente removido durante o processo de extração, e a lavagem deve ser suficiente para que as paredes ao redor do tubo possam ser lavadas.Além disso, o esvaziamento e sopro do tubo é uma etapa necessária, que reduzirá ainda mais o resíduo de matéria orgânica.
Para obter mais informações sobre a extração de RNA, siga nosso site:
www.foreivd.com para mais informações.

Horário da postagem: 01 de dezembro de 2022